タイチンの PEVK ドメインがアクチンフィラメントに巻きついて弾性力を発揮するという ‘winding filament’ 仮説(タイチンは’winding filament’なのか?筋収縮における新たなねじれ)には一定の説得力があったものの,最新の観測技術はその仮説を否定しつつある.
武器となったのは原子間力顕微鏡と磁気ピンセットというナノメートルサイズの観測技術および操作技術が発展したことである.科学技術の発展が次の科学技術の発展を促すという指数関数的な技術発展の時代に我々はいる. “Technology will always win.” という名言を噛み締めている.

原子間力顕微鏡の探針
現在ではタイチン分子の折りたたみ(フォールディング)と伸展(アンフォールディング)が弾性エネルギーの根源ではないかと考えられており, titin folding 仮説という.今回はその根拠となる論文を紹介する.
正直なところ,俺は化学や物理学の専門家ではない.英語論文を和訳するにあたり,専門用語のオンパレードに萎縮しがちである.この分野の英和辞典が欲しいと切実に願っている.
The work of titin protein folding as a major driver in muscle contraction Annu Rev Physiol. 2018 February 10; 80: 327–351. doi:10.1146/annurev-physiol-021317-121254.
Abstract
Single molecule atomic force microscopy and magnetic tweezers experiments have demonstrated that titin Ig domains are capable of folding against a pulling force, generating mechanical work which exceeds that produced by a myosin motor. We hypothesize that upon muscle activation, formation of actomyosin crossbridges reduces the force on titin causing entropic recoil of the titin polymer and triggering the folding of the titin Ig domains. In the physiological force range of 4–15 pN under which titin operates in muscle, the folding contraction of a single Ig domain can generate 200% of the work of entropic recoil, and occurs at forces which exceed the maximum stalling force of single myosin motors. Thus titin operates like a mechanical battery storing elastic energy efficiently by unfolding Ig domains, and delivering the charge back by folding when the motors are activated during a contraction. We advance the hypothesis that titin folding and myosin activation act as inextricable partners during muscle contraction.
Keywords
Muscle contraction; titin; protein folding; polymer physics; single molecule; force spectroscopy
要約
単分子原子間力顕微鏡および磁気ピンセット実験が明らかにしたのは,タイチン Ig ドメインが牽引力に対抗することができるということであり,それはミオシンモーターにより産生される仕事を超える機械的仕事を発生させる.我々は筋活動を仮定した,アクチン-ミオシン架橋形成がタイチンポリマーのエントロピー反動を引き起こし,タイチン Ig ドメインの折りたたみを惹起することでタイチンにかかる力を減少させると.筋肉内でタイチンが動作する状況下での 4-15 pN という生理学的な力の範囲では,単一の Ig ドメインの収縮の折りたたみはエントロピー反動の 200 % の仕事を発生させることができ,単一のミオシンモーターを失速させる最大の力を超える力を発生させることができる.ゆえにタイチンは機械式バッテリーのように動作し, Ig ドメインを伸展することで弾性エネルギーを効率的に蓄積し,収縮期にモーターが活性化した際に折りたたみにより電荷を再充電する.我々はこの仮説を発展させた.つまり, titin folding およびミオシン活性化は筋収縮において不可分のパートナーとしてふるまうのであると.
Introduction
前世紀のほとんどの期間,人体における最大の蛋白質であるタイチンの存在は知られていなかった (1). 驚くことではないが,筋収縮の理論は当時知られていた分子であるアクチンとミオシンに基づいていた.しかし,フィラメント滑走説の発見の前夜には,いくつか競合する仮説が存在しており,不思議なことに,筋収縮におけるタイチンフィラメントのような役割を予言したものもあった.1952 年,物理化学者の Henry Erying と親しかった研究者の Milton Polissar は運動速度理論を用いて 100 個の独立した収縮単位からなる分節状フィラメントの存在を示唆しており,その収縮単位は力の存在下に機械的仕事をなして短縮することができるというものであった (2,3,4,5). 彼のアイデアは,フィラメント滑走説が十分な実験結果 (6,7,8,9) を伴って発展してきた時には道の端に落ちてしまった.魅惑的な運命のねじれにより,我々はこんにち,両方の仮説が正しく,両方の機序が筋収縮に寄与することを知っている.今や,次の点を確立する決定的な根拠が存在する.つまり,筋サルコメアは三つの主要なフィラメント,つまりタイチン,アクチンおよびミオシンからなり,共に弾性エネルギーを蓄積し,機械的仕事を実行するために働く (10). 筋肉における三番目のフィラメントとしてのタイチンの根拠は,最初は疑いの目で採用され (11), サルコメア内部のタイチンの存在が広く受け入れられるまでには数十年を要した (12,13). ミオシンの発見とフィラメント滑走説 (14) の発展の間で一世紀近くが経過し,我々は間違いなくタイチンの生理学的機能の理解の初期段階に達したばかりである.
タイチンは人体において最大級の蛋白質であり, 370 万ダルトン (15) と既知のアイソフォームで最大の分子量を持つ単一のポリペプチド鎖からなる.各タイチンフィラメントは半サルコメア全体にわたり,それらは 4 つの異なる領域に分割され,顕微鏡で容易に認識できる (Figure 1A). 筋肉の I-band は長さが可変で主にアクチンフィラメントからなり,タイチンと平行に走行している.サルコメアの緊張に依存して,アクチフィラメントのある部位が A-band 内部に向かって走行する.その A-band は太いフィラメント末端により区画されており,太いフィラメントとは 294 個のミオシン分子の束のことであるが,多くの脊椎動物において 1.65 µm の長さで見出される (16). 筋肉の横断面を電子顕微鏡で観察すると,線維が六角格子状に配列しており, 6 本のアクチンフィラメントが各ミオシンの束の周囲を取り囲んでいるのが観察できる (17). 太いフィラメントは M-line 内の蛋白質で互いに固定されており,一方アクチンフィラメントは Z 盤内のアルファアクチニンにより係留されている.
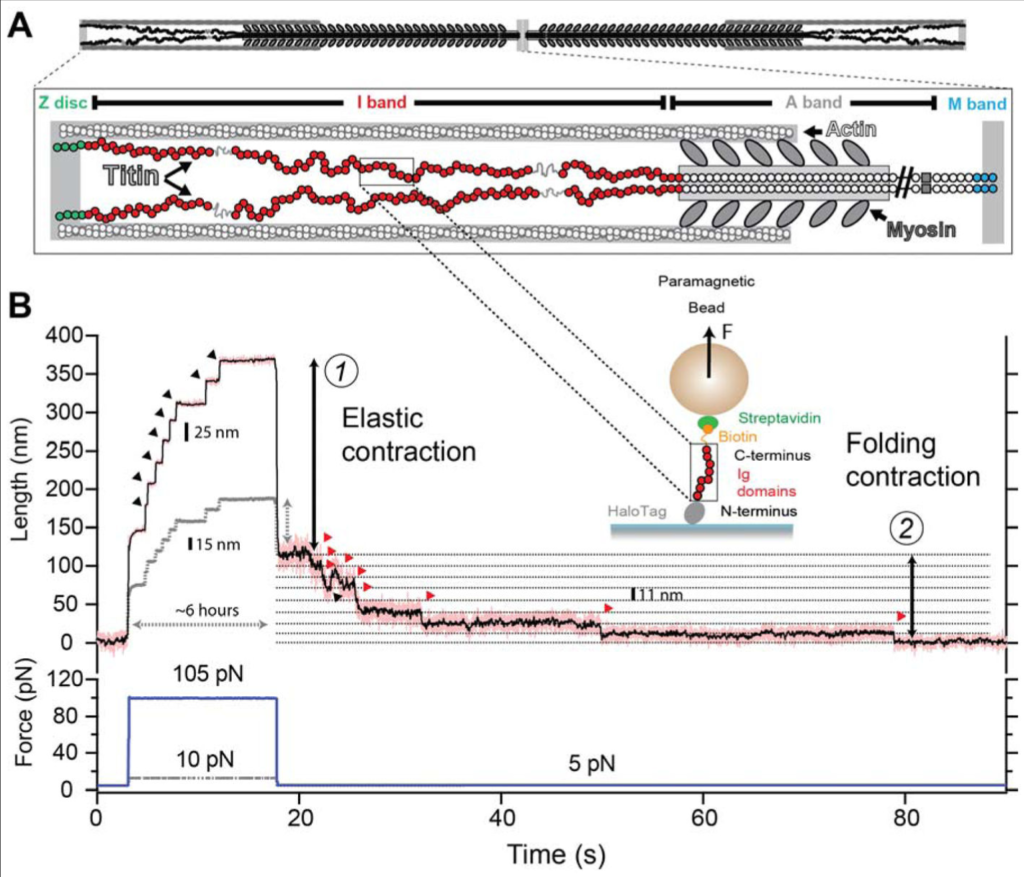
タイチンは筋肉のこれら 4 つの領域で容易に見つかる.タイチンの N 末端は 9 つの免疫グロブリン (Ig) ドメインからなり (Figure 1A, 緑色), Z 盤に蛋白質で結合している.I-band 領域のタイチン (Figure 1A, 赤) は選択的にスプライスされたエクソンからなり, 100 個にも及ぶ Ig ドメインをコードしており,本質的に無秩序な領域と同様で,いわゆる N2B 配列および PEVK 配列と呼ばれ,プロリン,グルタミン酸,バリンおよびリジン残基に富む.特記すべき重要なことであるが,選択的スプライシングにより I-band で見つかる Ig ドメインおよび非構造的アミノ酸の数は,各筋肉群により異なると考えられている.最長の骨格筋アイソフォームは 100 個の Ig ドメインを有するのに,心筋組織で支配的な N2B タイチンのアイソフォームはたった 47 個の Ig ドメインと 735 個の非構造アミノ酸残基を有するに過ぎない.タイチンのアイソフォームの大きさは,いかなる所与のサルコメア長でタイチンが力を経験するかを理解するために重要である (18). タイチンの A-band 領域は (Figure 1A, 灰色) Ig ドメインおよびフィブロネクチンドメインからなり,それらは太いフィラメントに固く結合している.最後に,タイチンの M-band 部位 (Figure 1A, 青) はタイチン偽キナーゼ (19,20) および 10 個の Ig ドメインからなり,隣接するタイチンおよび他の構造蛋白質を結合している.このレビューは主にタイチンの弾性 I-band 切片の力の発生能力に関係しているが,タイチンと筋肉の微細構造を議論している優秀な資源は多数存在する (21,22,23,24,25).
筋収縮のフィラメント滑走説においては, ATP 加水分解と無機リン酸の放出がミオシン頸部の構造変化をもたらし,ミオシン頭部がアクチンに結合する.この動作が太いフィラメントを動かし,アクチンに対する力を発生させる (9). 筋収縮の際にミオシンが単独でアクチンフィラメントを滑走させ, ATP の化学エネルギーがミオシンモーター周期にエネルギーを供給するという点は間違いない (26,27,28,29) が,短縮時のタイチンの役割はほとんど理解されておらず,明確にするために持続的な焦点を当てることが筋肉コミュニティに求められていた.タイチンがいかにして筋収縮の際に力を発生するのかについては,いくつか競合する仮説が存在する (22,30,31,32) が,筋線維レベルで分子機構を解明する試みは失敗してきた.過去 20 年間にわたり,タンパク質工学と組み合わせた分子間力分光法における進歩により,力の下でのタイチンの力学の研究が進んできた (10,33,34,35,36,37,38,39). 分子間力分光法のデータの示すところは,タイチンは Ig ドメイン分子の多くの腕を伸展したり再度折りたたんだりすることで弾性エネルギーのほとんどを蓄積し,提供するというものである.今や我々の責務は,タイチン折りたたみの力学をフィラメント滑走説の枠組みに組み込むことで,筋収縮の知見を発展させることである.さあ,やってみよう.
Studies of titin under force
化学的・熱的変性などの伝統的な過半数の生化学技術は筋肉内のタイチンの機能を理解するには実用的ではない. 生体内でタイチンは 15 pN までの機械的負荷のもとで動作するが,それは Z 盤への付着と太いフィラメント先端との間のベクトルに沿って適用される負荷である.原子間力顕微鏡 (AFM) や磁気ピンセットおよび光ピンセットなどの単分子間力分光法技術は,生体内で経験するのと同じ協調反応を通じてタイチンのナノスケールの機序を掘り下げ,過半数の技術よりもっと生理学的な洞察をもたらしてくれる.蛋白質レベルのコンフォメーション変化を引き起こす細胞プロセスの多様性を修飾する力の新しい観点により,これらの手法は急速に細胞の機序を理解するための不可欠の技術となりつつある.
蛋白質分子間力分光法の実験の実施には,固定表面と顕微鏡プローブの間の単一のポリペプチド一本鎖を必要とし,そのプローブは張力をかけられるよう配置できるものである.AFM 研究のために,張力プローブは窒化ケイ素製のマイクロカンチレバーであり,蛋白質末端に共有結合でも非共有結合でも取り付けることができる (40,41). 磁気ピンセットについては,磁界を受けた磁性体粒子を付着させた蛋白質を一本鎖に引き伸ばす (10,40,42,43). 光ピンセットはレーザートラップを用いるもので,誘電体粒子をポリペプチドに付着させ力を適用する (44,45). すべての例で,張力プローブの位置は高速レーザー検出器またはビデオ顕微鏡でナノメーター,または最低でもオングストローム精度で計測される.伸展によるポリペプチド長の漸進的な変化,またはドメインのアンフォールディングおよびフォールディングによる突然の変化は高い時空間解像度で計測可能である (46,47,48,49).
初期の単分子間力分光法の実験では, DNA (50,51) と多糖類 (52,53) の機序,受容体とリガンドの解離 (54), モーター蛋白力学 (26,44), そして酵素のコンフォメーション変化 (55) を調査した.しかし,ごく初期のナノメートル解像度での可逆的な蛋白質のアンフォールディング記録は,マイクロメートル長のタイチンを基質として実行された (37). AFM を用いて,ウシの組織から精製された天然のタイチンは高い力で伸展され,カンチレバー先端を引き上げ面に戻すことで再フォールドできるようにした.タイチンはこれらの実験のための自然の選択であった.というのは,タイチンは多くのドメインがほぼ同じ大きさの免疫グロブリンからなり,一定速度で単分子を牽引する実験において特徴的な「鋸歯状」パターンを呈していたからである.同様の結果が光ピンセットを用いて天然のタイチンフィラメントを高い力で伸展する実験で示されており,蛋白質の段階的様式または漸増様式が得られ,これは単一ドメインのアンフォールディングが起きたことを示唆している (35,39). 遺伝子組換え蛋白質工学および分子クローニング技術は,非常に詳細に研究された弾性タイチン I-band の位置をより小さく,より制御可能にしている (33,56). これにより Ig ドメイン内部の機械的階層が明らかになり,近位 Ig ドメイン(N 末端に近い部位)の機械的安定性が I-band の遠位 Ig ドメインよりも低いことが分かった.さらに, HaloTag アンカーの蛋白質の N または C 末端への共有結合の工学は,強い力で長時間の研究のための遺伝子組み換えタイチンの特異的固定化への道を開いた (41,49,57).
過去 20 年間にわたり,分子間力分光法は計装と技術において大きな改善の恩恵を受けた.電位固定法を鑑みるに,単一のイオンチャネルゲートの実験の初めての成功を支えたものであるが, AFM のための原子間力固定法の発展は蛋白質フォールディングのエネルギー論について我々の理解を大きく進歩させた (34,38,58). 原子間力固定分光法は明白に,定義された牽引力の下での,単一の蛋白質ペプチドのアンフォールディングおよびフォールディングの軌跡に付き添い,オングストローム単位の解像度で蛋白質の末端から末端までの長さを追跡する.これにより,力学と外力依存性の蛋白質フォールディングや他の生化学反応との研究が進んだ (59,60). もっと最近では,受動的に無限の帯域幅で動作する蛋白質磁気ピンセットの発展により, 15 pN 以下の力でタイチン Ig ドメインにおけるフォールディング・アンフォールディング遷移平衡を直接観測できるようになった (10,43). この種の実験は,生体内で動作するのと極めてよく似た状態でのタイチンを探索するための自然なシナリオを提供してくれる. Figure 1B に実験例を図示したが,今日我々がタイチンの機序について知っていることの多くを要約してある.この実験においては 8 本の腕を持つ単一の蛋白質 Ig 91 (I91) ドメインが磁気ピンセット装置の中で一定の伸展力の下に固定されている.重要なことなので書いておくが,この I91 ドメインは古い文献 (36,61) で言及されている I27 ドメインと同一であり,命名法が変化したのみである.その蛋白質は当初, 105 pN もの非生理的な強い力であっという間にすべての Ig ドメインが伸展してしまった.これは最初に弾性の伸展が起こり,引き続いてアンフォールディングと 8 つの Ig ドメインの伸長にはたった 20 秒間しかかからなかった.対照的に, 10 pN の生理的な力では I91 のアンフォールディングは数時間を要した (Figure 1B 灰色点線). ポリペプチドにかかる力が 5 pN にまで減少すると,タイチンは即時弾力コイルに変化して引き続き段階的フォールディング反応を示した.
原子間力分光法によりアンフォールドした最初の蛋白質がタイチンであったため,引き続いて蛋白質フォールディング物理学を理解するモデルとなった.タイチンのように球状に折りたたまれることの機械的安定性は静電相互作用,水素結合および疎水相互作用に依存し,蛋白質の配列および位相により支配される. Figure 2A に, 6 pN の機械的力で可逆的にアンフォールディングするタイチン Ig ドメインの詳細図を示す.力の大部分は最初の (A-A’) と最後の (G) ベータ鎖との間の水素結合により維持され (62), 偶発的にドメインの完全なアンフォールディングが起きる.これがタイチン Ig ドメインにとってアンフォールディングが起きる機序と考えられており, I91 との高い構造的類似性が認められる.一旦アンフォールディングが起きると,そのポリペプチドはランダムコイルとなる.その意味するところは,末端間の距離は,よく知られているポリマー物理学法則による張力と規模により定義されるということである (63). 例えば, 6 pN において,折りたたまれた I91 ドメインは末端から末端への長さが約 4 nm であり(原子座標から推測.詳細は PDB 1WAA 参照のこと(64)),一方で伸展されたポリマーは 17 nm の長さを持つ.ゆえに,フォールディングとアンフォールディング遷移期に観察されたステップは 6 pN の力の下では 13 nm のサイズを有する. Figure 1B もまた 5.0 pN, 10.0 pN および 105 pN の力でのこの大きさの変化を示している.フォールディングおよびアンフォールディング遷移をスキャンすると広範囲の力が自由結合鎖モデルに適合するデータを許容することが分かる (63). その輪郭長は 25 nm であり, Kuhn 長は 0.9 nm である (Figure 2B, 実線). ゴムバンドの線形弾性とは対照的に,ポリマーのエントロピー反動は高度に非線形性であり,生理学的な力の範囲を超えてその最大伸展の 50 % にも達する.この高い力においては,蛋白質のアンフォールディングのステップサイズは漸近的に輪郭長に近づく.
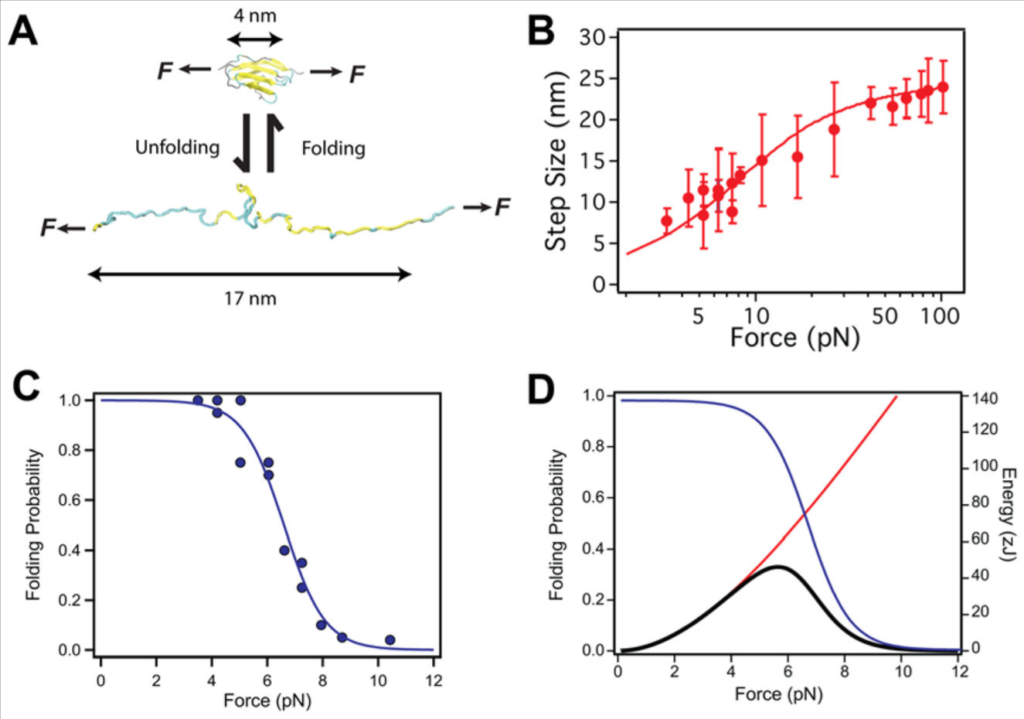
Ig ドメインのフォールディングによって発生した機械的仕事は,フォースクランプ磁気ピンセットを用いて定量可能である
(A) 機械的張力の下での Ig ドメインのアンフォールディング,再フォールディング. 6 pN までの牽引力の下では, Ig ドメインは,ポリマー物理学のスケーリング則に従い,平衡状態でアンフォールディングおよび再フォールディングし,約 13 nm 伸長または短縮する. (B) アンフォールディング・再フォールディングの間に I10 および I91 のタイチン Ig ドメインの力の関数として測定されたステップサイズの大きさ.ステップサイズの依存性は, Kuhn 長 lk = 0.9 nm, 輪郭長 Lc = 25 nm の自由結合鎖モデル(実線)でよく記述できる. (C) 所与の牽引力で,折り畳まれたドメインの平衡分画(青い円)から計算された, Ig ドメインのフォールディング確率.タイチン Ig ドメインは, 4 pN の低い力でアンフォールディングし, 10 pN の高い力でフォールディングすることができる.そのデータは, 6 pN までの中間点を有するシグモイド関数(実線)によってよく表すことができる. (D) タイチン Igドメインのフォールディングにより生成される予想される仕事は,フォールディング確率(青線)と、フォールディング収縮によって行われた仕事(赤線)との積として得られ,ステップサイズに適合するよう用いた自由結合鎖モデルの数値積分から得られる.タイチン Ig ドメインは 5.7 pN で 46 zJ のピーク仕事を生成し,それはミオシンモーターにより供給されるエネルギーに匹敵する.
タイチン Ig ドメインのそれぞれがアンフォールディングする際の率は大幅に異なる.そのため生理的な伸展中には,全タイチン Ig ドメインの一部のみが伸展される可能性がある. I-band から採取した 8 本腕の Ig ドメインの野生型セグメントをクローニングしてみると,タイチン内部の機械的階層が明らかになった.近位 I-band 内部の Ig ドメインは Z 盤に近いほど,遠位 I-band Ig ドメインよりもはるかに小さい力で伸展し,太いフィラメントに近い (36). ゆえに, I91 が 10 pN で伸展するのに数時間を要するにもかかわらず (43,65), 近位 Ig ドメイン 10 (I10) ははるかに早く伸展する.I10 での磁気ピンセット実験で明らかになったのは, 6.0 pN の力では, 20 分間で約 66 回のアンフォールディングイベントが発生し,それぞれのアンフォールディングイベント間には 15 秒間があるということである.それゆえ 10 pN においては, I10 は数秒間で伸展すると推測されるが,これは実験で検証する必要がある.野生型の I4-I11 セグメントを用いた実験で 5.0-6.6 pN 間の力では,素早い伸展イベントが観測され,それゆえ機械的安定性は低いとされた.これは近位 Ig ドメインが生理的な力の範囲で容易に伸展するという視点を支持している (10).
タイチンドメインの伸展経路に関しては豊富な情報があるにもかかわらず,折りたたみ経路の観察はさらに見つかりにくく, force clamp 法のような技術の発展を待たねばならなかった (58). その force clamp 法により,ランダムコイルから自然な折りたたみへの蛋白質の形態の変形の間に,三つの異なる遷移があることが明らかになった (34). その結果,従来の一括法を用いて見られたのとは全く異なる図が描かれることとなった.最初に,ある蛋白質は力の変化に対応して力の下で弾性収縮力を経験して伸展する. Figure 1B を参照されたい.力が減弱するとポリマー内のエントロピーが大幅に増大し,蛋白質が折りたたまれるようにするに違いない.このエントロピーを除去するにはエネルギー障壁を横断する必要があり,その障壁はエントロピー障壁として知られる牽引力により作られる (66). この遷移は,モルテン・グロビュール遷移への折りたたみとして知られており,疎水性残基の蛋白質コアへの隔離により駆動され,その結果 Figure 1B に示すように下向きの段階となると考えられている.配置エントロピーのさらなる減少は,モルテン・グロビュールから自然状態への発展の過程を通じて達成される.それは機械的クランプにおける水素結合の形成を通じてなされるようである.このモルテン・グロビュールの自然遷移は蛋白質末端間の長さの微小変化を引き起こし,モルテン・グロビュールと自然状態との間で劇的に機械的安定性が異なることで,異なる状態が検出されることになる (67). 化学的変性や phi-value analysis の一括法の研究で想定されるこの古典的な「二状態」モデルは,伸展され折りたたまれたモルテン・グロビュールや,単分子フォールディング実験で観測される自然状態とは相容れない.単分子原子間力分光法は,これら従来の手法よりも,このフォールディング経路についてより多くの詳細をもたらしてくれる.
Protein folding performs mechanical work
Force-clamp 原子間力顕微鏡の発明以前には,力の下でのフォールディングの計測は非常に単純なものしかなかった (33). これらの実験では,八つの腕のタイチン Ig ドメインを含む蛋白質の構成が,一定速度での AFM カンチレバーの牽引による最初のアンフォールディングとなった.引き続いてカンチレバー先端が試料表面からの力でごく短い距離(数十ナノメートル)下がり,接触なしにごく短時間そこにとどまる.この過程が蛋白質の residual force の残留効果である.カンチレバーが再度牽引されることで,その折りたたまれたドメイン数が計測できる.それらの実験から明らかになったのは,再フォールディング率がアンフォールドしている蛋白質の弛緩の程度に強く依存するということであり (33), 再フォールディング率は residual force に急激に依存するとのエビデンスが得られた.しかし,それら初期の実験においては,蛋白質がフォールディングする力の下では,直接計測ができなかった.Force-clamp AFM の発展は,世界で初めて機械的な力の下での蛋白質フォールディングの完全で鋭い直接観察をもたらしてくれた (34). ユビキチンドメイン腕からなる蛋白質構成を用いて, アンフォールドした蛋白質を牽引する力を「急停止」すると,その蛋白質はまず弾性収縮し,次いで遅延の後,牽引力に抵抗して機械的仕事を行い,完全に折りたたまれた長さに短縮することを Fernandez と Li が証明した.力の急停止後,続いて高い力のパルスが折りたたまれたドメインの指紋を明らかにし,これらの折りたたみの軌跡は,フォールディング過程が真実の観察であることを教えてくれる.より短時間の急停止での似た実験では機械的に脆弱な状態の存在が明らかになった.そこでは,完全に折りたたまれた蛋白質の前駆体として保存され,モルテン・グロビュール状態をコピーした状況に似ている (67,68,69). これは力の下でのフォールディング物理学を明らかにしたもので,そこでは力の急停止に反応して蛋白質はまず伸長した状態から折りたたまれた状態に弾性収縮し,そのエントロピーを大幅に増加させる.その折りたたみ状態から,蛋白質はさらに収縮して段階様式で短縮し,低エントロピーのモルテン・グロビュール状態になる.このモルテン・グロビュール状態において蛋白質は「成熟」して機械的に自然な形態に移行し,水素結合のネットワークを再構築して機械的に安定した状態になる.このシナリオにおいては,大部分の折りたたみ収縮は折りたたみ状態からモルテン・グロビュール状態への蛋白質の動きとして発生するが,水素結合ネットワークの再構築は蛋白質の末端間の距離をそれほど変化させない (67). 磁気ピンセット法を用いて,I10 と I91 の折りたたみ軌道のフォールディングおよびアンフォールディングのステップサイズを 30 pN 未満の力で計測してみると (Figure 2B), モルテン・グロビュール状態のもっと完全な根拠が得られる (70). 詳細に調べられたのはこれらたった二つのドメインに過ぎないが, 4-10 pN の同じ範囲の力の中で,アンフォールディング中の劇的な機械的安定性の違いにもかかわらず,フォールディング遷移を経験しているように見える.おそらくこれは各 Ig ドメインが同数の残基を有しており,モルテン・グロビュール遷移への折りたたみの間,大まかに同量のエントロピーを放電するからに違いない.しかし,構成ループエントロピー (71) や点突然変異がアンフォールディング経路に影響する (72) ことが明らかになったため, Ig ドメイン配列の多様性が生理的範囲内でのフォールディング率の間での階層を形成する結果となったのであろう.これは実験的に検証されなければならないことだが,この点において根拠の示唆するところは,筋収縮時にタイチン Ig ドメインは狭い力の範囲で折りたたまれるということである.このフォールディング収縮が AFM で観察されると,持続的かつほとんど稀に段階を示すことが分かった (34). 対照的に,同じ実験で磁気ピンセット法を用いると,最初の弾性収縮後,フォールディング収縮時に明確な段階(的収縮)を見せた (10,43,49). AFM と磁気ピンセット法でのフォールディングの軌道の形が異なる理由は,すべての単分子原子間力分光法の実験において,ポリペプチドの力学がテザー依存性であるということである (73). そのポリペプチドは探針が接触するほど速く動くことができるのみで,牽引する方向の鎖の力学は,探針と同じ有効な拡散係数を有する.三角形のカンチレバーは通常単分子の実験で用いられ,長さと幅がそれぞれ 310 µm および 20 µm であり,拡散係数は 1,200 nm2 と測定されている (73). カンチレバーの拡散係数を用いたフォールディングのブラウン運動力学シミュレーションはフォールディング軌道の再現性を可能とし, force-clamp AFM を用いたものと近似していた (66,74). 対照的に,直径 2.8 µm の自由浮動球体粒子は 175,000 nm2 までの拡散係数を持つ.磁気ビーズが試料表面と近接していてその値を若干減弱するにもかかわらずである (75,76). これは磁気ピンセットで見られる折りたたみ軌道の違いに寄与している.ここで注意が必要である.美しい段階的フォールディング軌道が磁気ピンセット法で観測され,フォールディングエネルギーの風景 (77) およびフォールディングの仕事 (10) の画像がより良い解像度で得られたからと言って,それが生体内でのタイチン蛋白質のフォールディングの真実の拡散のふるまいを表しているわけではない.タイチンが Z 盤と M-line という巨大な分子構造に係留されていることから,また,シャペロンを含む混雑した環境で動作することから,生体内でのフォールディング収縮は AFM で観測されるような滑らかなフォールディング収縮のようにもっと拡散の制約されたフォールディングのふるまいに似ている可能性がある (34,74).
我々は提案する.正常な筋活動時のタイチンへの負荷の生理的な力の範囲とは,折りたたみと伸展の平衡状態で Ig ドメインが存在する範囲を超える力の範囲であると.これはフォールディング確率を通じて定量される可能性があり,タイチン Ig ドメインが任意の力で折りたたまれる時間の割合を計測するものである.タイチン Ig ドメイン I10 および I91 のフォールディング確率を磁気ピンセットの原子間力分光法で計測し, Figure 2C (青丸)に要約してあり,シグモイド関数に適合しており,フォールディング反応の midpoint と steepness を記述してある (青線, Figure 2C, Figure 2D). タイチン Ig ドメインが 4 pN 以下の力で完全に折りたたまれ, 10 pN より大きい力では折りたたまれないことは明らかである.現在ではこの力の範囲は 15 pN まで拡大しているとの根拠がある.タイチンのこの I-band 領域はシステインが豊富で (78,79), これらの多くの残基はジスルフィド結合を形成し,ポリマー力学をアンフォールディング状態に変える.その結果,タイチン Ig ドメインが 15 pN までのより高い力でアンフォールドしやすくなり,折りたたみ状態における機械的安定性をより失わせやすくする効果がある (80).
Force-clamp 分光法が付加的に可能にした単純な定量化として,タイチンドメインが適用された力に抗してフォールディングして実行する収縮の仕事量がある.いかなる力を与えても,システム上またはシステムによりなされる収縮の仕事量は,力とステップサイズの積である. I10 および I91 のフォールディングによりなされる仕事は Figure 2D の赤い曲線で示したが,力に対応する自由結合鎖曲線の積分である.フォールディング確率でなされた収縮仕事を代表する乗算曲線は,与えられた力での蛋白質フォールディングによるエネルギー回復の期待値を与えてくれる (黒の曲線, Figure 2D). この分布が示すところは,タイチン Ig フォールディングが, 5.7 pN の負荷に対して 47 zJ と期待される最大の仕事を発生させるということである.10 pN に近い高い負荷では,エネルギー回復は最大化するが,ドメインの折りたたみ頻度は減少する.代わりに,軽い負荷においてはエネルギー回復は最小化する.というのは,蛋白質フォールディングのステップサイズが小さく,ほとんどのエネルギーが弾性収縮で消費されてしまうからである.ゆえに我々は,生体内で単一のタイチンフィラメントにかかる力を定義できれば,タイチンフォールディングを通じて蓄積され解放されるエネルギー量を予測できる.
How the three filaments work together
単分子の実験は,蛋白質フォールディングのエネルギー理論について理解するのみならず,単一のミオシンモーターによる力の発生の基礎について理解するための強固な基盤を提供してきた.これはタイチン Ig フォールディングの収縮特性とミオシンパワーストロークとの間の公平な比較を意味している.ミオシンモーターの独奏または合奏における単分子原子間力分光法は, ATP で駆動されるモーターが実行できる利用可能な仕事量に固有の制限があることを明らかにした.仮に十分に大きな負荷がモーターにかかると,そのモーター出力はゼロに低下してしまう.自転車で坂道を登る時,物理学のこの一かけらを痛感させられる.初期の筋肉のパイオニアである A. V. Hill はこの効果を捉えて彼の有名な「Hill の公式」とした.つまり,収縮速度と負荷との間の双曲線関係を記述したものである (81). 試験管内の実験が明らかにしたところでは,免荷状態の筋肉の速度は極めて速いが,少しでも負荷をかけると, II 型ミオシンモーターの滑走速度が急激に低下してしまう.骨格筋のミオシンの機序を探索する精密な実験で明らかになったのは,等尺性状態下でモーター 1 個が産生する力の平均値は 3.4 pN である (26). まれに,このミオシンが持続的に発生させる力が 6-7 pN の範囲に遷移することがある.さらに最近では,単分子の力-速度関係を定義することを目的として,同じ実験が 8 個までのミオシンモーターを束にして実施された (Figure 3A, 緑色曲線) (82). このミオシンモーターはやはり 6.0 pN の負荷を適用するとアクチンフィラメントの変位が正味ゼロになってしまい,この分子マシンの実行には上限値が存在することを再確認し,それは高解像度光ピンセット法でも再確認された (29). 他にキネシン, V 型ミオシン,ダイニンの失速する力はすべて 6-7 pN の範囲に収まっていることが示されている (83,84,85). これらの実験は force clamp 法を用いて変換器の応答を減弱させ,分子モーターの動力発生を最大化している.これらのデータから論理的に導かれる結果とは, 6 pN の力では II 型ミオシンモーターはアクチンフィラメントに収縮の仕事を発生させることができないということである.対照的に,タイチンのフォールディングが 6 pN の負荷においてピークパワーを提供することを我々は示したが,そこでは II 型ミオシンモーターは失速していた (Figure 2D, Figure 3A) (10).

タイチンは,筋肉がミオシンの失速する力を超える負荷で働くことを可能にする
(A) 単分子研究からのミオシンモーターとタイチン Ig ドメインのエネルギー論の比較: 8 個までのミオシン頭部が光トラップ中のアクチンフィラメントを推進した実験からのデータは,ミオシンが 6 pN の負荷に対して失速していることを示している (82). そのデータは A.V.Hill の公式(緑色の曲線)に合致する.あるいは,タイチン Ig ドメインは 5.7 pN まで(青色曲線のピーク)の力で 46 zJ の仕事の最適な供給を達成する.したがって, 6-12 pN (灰色の陰影部分)の負荷では,ミオシンの出力がマイナスであるのに対して,タイチンフォールディングは有用な収縮の仕事を発生させることができる. (B) タイチンフォールディングによってミオシンモーターが前進する: (1) 筋肉が活性化される前にタイチンは 12 pN の受動的負荷全体を負担し,その結果, Ig ドメインのアンフォールディングが起こる. (2) カルシウムが放出されると,アクトミオシン架橋が形成されるが,そのモーターは失速しているため, 6 pN の力しか発生させることができない.しかし,モーターによって発生した力はタイチンドメインが受ける負荷を 6 pN に減少させ,結果として 10 nm までの再フォールディングステップをもたらす. (3) 蛋白質フォールディングのプロセスはタイチンフィラメントを短縮し,ミオシンモーターの負担を軽減してパワーストロークの完了を可能にする.このメカニズムから,タイチンは架橋形成なしでフォールディングすることができないだけでなく,パワーストロークの完了もまたタイチンフォールディングに依存することも明らかである.従って,タイミンとミオシンは、高い負荷に対してサルコメアを短縮するために手を携えて働く.
筋肉収縮時にこれが発生する際のもっともらしいシナリオは,各タイチンフィラメントが 12 pN の力を支えるために,いつサルコメアが受動的に負荷されるかである (86,87). 筋肉の活動前後においてタイチンとミオシン間で,どうやってこの負荷を分配しているのだろう?仮に 147 本の双頭のミオシン分子が半分の太いフィラメント内にあり,またサルコメア半分あたり 6 個のタイチン分子があると考えるなら,タイチンフィラメントあたりのミオシン分子の絶対的な最大比は 25 である.しかし,筋短縮中にすべてのミオシン頭部がアクチン結合部位に結合しているわけではない.筋ミオシンは非連続モーターであり,収縮中にミオシン頭部がアクチンに結合するパーセンテージは結合部位に結合可能な最大数の 10-20 % 程度と推定されてきた (88). 収縮中の筋線維の追従を計測することで,1 本の太い線維あたり 84 個 (~ 24 %) までのミオシン頭部が従事している可能性があると計算され,結合した頭部の数が急速にフィラメントのオーバーラップおよび短縮速度の両者を変化させていた (89). しかし,タイチンの弾力性はこの実験では考慮されておらず,そのドメインが折りたたまれてタイチンは硬化しているとして,収縮している筋肉で従事しているミオシンモーターの数を過剰推定する結果となった.ウサギの大腰筋の 3.1 µm やヒト伸展筋の 3.4 µm など生理的なサルコメアの伸展では,太いフィラメントと細いフィラメントのオーバーラップの程度が減少するとミオシン頭部の結合数が少なくなる (10,90). ゆえに,そのような伸展された長さでは,収縮を支えるタイチン分子 1 個あたりのミオシン頭部の数はたった 1-2 個に過ぎないと計算された.
仮説のシナリオに戻るが,タイチンは筋収縮に先立って 12 pN の負荷全体を支え,近位 Ig ドメインを動員してアンフォールド状態にする (Figure 3B, 上部パネル). 例えば野球の投手のワインドアップ中のように,これは筋肉の爆発的収縮に先立って起きる可能性がある.筋肉の活動後の数ミリ秒間で,最大数の架橋が形成されてサルコメア内の三つの筋フィラメントからなる機械的ネットワークに負荷が分配される.ミオシンモーターは最大 6 pN の動力を発生させるが,サルコメアを短縮させるほど十分ではなく,全てのタイチン Ig ドメインにかかる負荷を 6 pN に減弱させる (Figure 3B, 中部パネル). この結果まず非常に急速に全ての伸展されたタイチン分子がエントロピー反動を起こし,続いてタイチンのフォールディングが起きて最大可能な仕事をなして力を回復させる (Figure 3A, 青曲線ピーク). 従来の仮説ではタイチンはエントロピー反動経由でのみエネルギーを回復するとみなされていたが,そうではなく,ここで回復するエネルギーの大半は実際にはフォールディング収縮に由来しているのである. 12 pN の力において,折りたたまれた状態からランダムコイルに行くことで達成される輪郭長の変化は 15.8 nm である. 6 pN までの力を減弱させるにおいて, 5.9 nm のエントロピー反動に引き続いて 9.9 nm のフォールディング収縮が起きる (Figure 3B, 中部パネル). 蛋白質フォールディングは,伸展されたポリペプチドからの回復によるエネルギーの 63 % を含む一方,弾性収縮は 37 % を提供するに過ぎない.フォールディング収縮こそ,生理学的な力の範囲でのタイチンの弾性およびエネルギー蓄積の主要な要素に他ならない.ゆえに,このシナリオでは, 12 pN 以下の負荷ではフォールディングした Ig ドメインが失速したモーターを解放し,サルコメアには伸長するよりむしろ収縮することを許す (Figure 3B, 下部パネル). 12 pN でタイチンを伸展させることで蓄積されるエネルギーは,モーターが従事する時にのみ,戻ってくる.この素晴らしいメカニズムは, II 型ミオシンモーターが失速した力を超える負荷でも,サルコメアが動力を提供する可能性を容易に拡張するものである.
Titin folding dictates the optimal sarcomere length of a contracting muscle
タイチンフォールディング収縮の存在は,筋生理学の教義の再訪を保証する可能性がある.例えば,次のことは一般に受け入れられている.つまり,下肢の active force とサルコメア長との関係は太いフィラメントと細いフォラメントのオーバーラップの減少による,ということである.しかし,ヒト骨格筋においてサルコメア長 1.7-2.6 µm で上肢における力は既に最大に近い (91). 太いフィラメントはせいぜい 1.6 µm までの長さであり,そのため上肢のこの部位において減少した力は太いフィラメントの Z 盤に対する圧縮ではなさそうである.別の説明として可能性があるのは,この部位での等尺性の力が減少するのは全てのタイチン Ig ドメインが折りたたまれたままであるから,というものがある.ゆえに,タイチンポリペプチドの疎水性崩壊により発生する付加的な収縮力というものは存在しない.仮説的な骨格筋を考えてみよう.タイチンアイソフォームが中間的な長さと硬度で存在するとする (Figure 4A, 青曲線). このタイチンアイソフォームは筋肉からの最高のパフォーマンスを可能とする.というのは,タイチンフォールディング確率における最大の変化は,最大の active force 発生する部位内でまっすぐに落ち込むからである (Figure 4A, ピンク領域). グレー領域での最大等尺力の計測ではタイチンフォールディングの関与を欠いている.というのは,該当するサルコメア長でのタイチンはフォールディングにもアンフォールディングにもいずれの状態もありうるからである (Figure 4A, グレー領域). これらの一見単純な実験は筋生理学の基礎を支持しており,タイチンフォールディングの文脈で考慮すると,そう簡単なものではない.
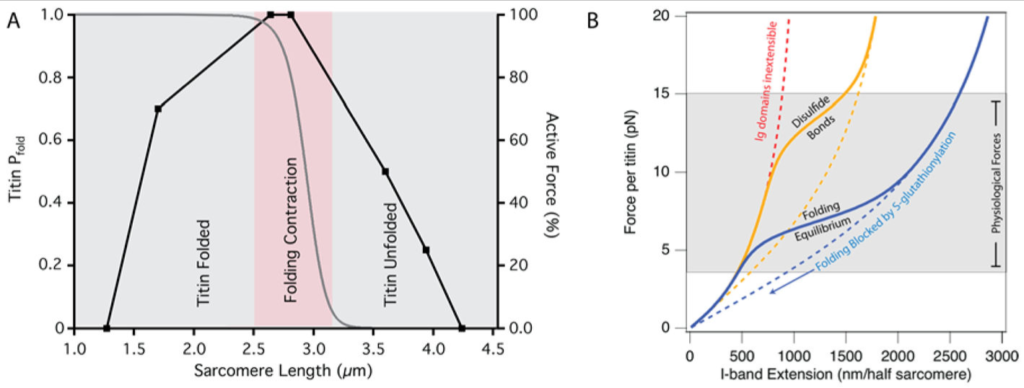
タイチンフォールディングは生理学的サルコメア長で生じる
(A) ヒトの骨格筋(黒い曲線)のアクティブフォース対サルコメア長の関係 (91) は,中間長のタイチンアイソフォームを有する仮想の筋肉について,タイチンのフォールディング確率とサルコメア長の関係(灰色の曲線)と重なる.ここで,アクティブフォースが最大となる範囲では,フォールディング確率が急激に 0 から 1 に変化し,筋収縮力が大きく増大する.アクティブフォース曲線の上昇局面では,タイチンは折りたたまれたままであり,約 2.5 μm 未満のサルコメア長で力の発生に寄与することができない.下行局面では,3.1 μm よりも大きいサルコメア長ではフォールディングがほとんどないため,タイチンは主にエントロピー反動復元力を提供する.すべての筋肉がアクティブフォース対サルコメア長の関係で動作するわけではないので,各筋肉群は,その特定の筋肉群の作動範囲に入るように,選択的にスプライスされたタイチンアイソフォームを有する可能性が高い (87). (B) タイチン I-band が多くの離散 Ig ドメインに分節化していることは,特別な材料特性を付与する.正常な生理的条件下(固体の青色の曲線)のタイチンは, 4-15 pN (灰色の陰影領域)の生理学的な力の範囲内でその Ig ドメインのフォールディングおよびアンフォールディングを経験する.フォールディング反応を完全に阻止することにより,タイチンのフォールディング確率,ひいては弾力特性を変えることができる(例.S-グルタチオニル化,青色の破線曲線).酸化的ストレスまたは酵素を介した分子内ジスルフィド結合の導入は,フォールディング反応が起こる力を増加させる(金色の曲線).小分子またはシャペロンの結合は,タイチンの機械的安定性を増加させ, Ig ドメインをより伸展不能にし,タイチンの全体的な剛性を増加させる(赤色曲線破線).実際に、タイチンの弾力性はこれらの曲線のすべての混合物であり,筋肉の代謝状態に依存する.
Why protein folding?
筋肉の可逆的変形は,おそらく他の組織とは比類のない材料特性である.筋肉は材料疲労やヒステリシスなしに伸展と弛緩の持続的なサイクルを経験しなくてはならない.いくつかの筋肉群では,完全収縮から完全伸展状態に至るとサルコメアの長さが 300 nm にまで伸展する可能性があり,これはサルコメア長の 10 % の増加にあたる (90,92,93). 可逆的な筋弾性に寄与するその蛋白質は,相反する二つの役割を満たさなくてはならない.つまり,筋肉は容易に変形でき,拮抗筋の収縮を妨害しない程度には柔軟でなくてはならないが,筋肉過伸展や筋収縮時に解放される膨大な弾性エネルギー蓄積による損傷を避ける程度には固くなくてはならない.ポリペプチドフィラメントは,正しく構造化されれば,弾性エネルギーを蓄積し解放するための優れた材料であることが分かった.
多くの細胞骨格構造は筋肉が動作する範囲の力では硬い.微小管はミリメートル以上の永続性の長さを有している一方で, f-アクチンは 10 ミクロンの永続性の長さを有しているのみである (94). ケラチンやビメンチンのような中間フィラメントは数百ナノメートルからマイクロメートルまでの永続性の長さを有する (95). I 型コラーゲンは三つの独立したポリペプチド鎖の三重らせんで構成されるが,数千ナノメートルもの永続性の長さを有すると考えられている (96). これらの素材のうちで有用な弾力性を蓄積するメカニズムを有するものは存在しない.理由は単純で,それらは硬すぎるのである.正常な筋肉の動作時に単一の蛋白質フィラメントが経験する力の範囲 (1-15 pN) を超えると,これらの蛋白質は既に完全に伸展されているが,硬い竿のように,筋肉の伸びに抵抗するだろう.代わりに,非構造的または伸展したポリペプチド鎖は多様な技術で特徴づけられてきたし, Kuhn 長の範囲は 0.8 nm から 1.2 mn であると示された(永続性の長さは 0.4 nm から 0.6 nm に等しい). 5 pN 以下の力では,あるランダムポリペプチドコイルはほぼ Hookean ばねとしてふるまうだろうが,より大きな力では,そのランダムコイルは非線形のふるまいを示し,伸展が増加するにつれてより急速に力を増す.最近の発見では,生理的な筋肉の活動時のタイチンは 4-15 pN の力の下では次の疑問を提示する.この力の範囲で,単純なランダムポリペプチドコイルよりも多くの弾性エネルギーを蓄積することができるような形態を,ある素材に取らせることは可能なのだろうか?
タイチンの構造はこの問題に対して,非構造セグメント (PEVK, N2B) および多くのタンデムからなる Ig ドメインの両者でもって特別な回答を提供してくれる. Ig ドメインへ分節化したタイチン I-band は筋肉の弾力性を生理的な力の範囲で大きく増加させる.そのポリペプチドは膨大なエネルギー (W = FΔL) を蓄積する.ここで F は FJC により定義される力であり L は観測される長さである.仮にタイチンが非構造ペプチドのみ含有すると仮定すると, 12 pN から 6 pN への力の減弱はその伸展長の 63 % の崩壊に由来することになる. 90 個までのアミノ酸を含有する非構造セグメントにとって,その長さが 15.7 nm から 9.9 nm に縮退することになり,その結果解放される弾性エネルギーは 34.8 zJ となる.今度は同じ 90 個のアミノ酸で分節化して折りたたむことのできるものを考えてみよう.12 から 6 pN に力が減弱すると,まず 34.8 zJ が放出され,次に付加的な 59.2 zJ がフォールディングに伴って放出される.エネルギー回復の合計は今や 94 zJ に増加し, 3 分の 2 はフォールディング収縮に由来することになる. Ig フォールディングとアンフォールディングはタイチンにダイナミックな適応を許してサルコメア長の幅広い範囲にわたる張力を提供するだけではなく,筋肉内のエネルギー蓄積と解放を増やすことにもなる.エネルギー蓄積と解放は,非常に精密な力の範囲内でのみ可能であり,そこでは蛋白質のフォールディングとアンフォールディングが起きている.我々は,フォールディング確率をまたいで,タイチンフィラメントの弾性ポテンシャルの蓄積を活用するような長さと力で筋肉が動作するよう調整されていると提案する.さらに,細胞シグナリングを通じてタイチンのフォールディングを操作することで,筋肉は弾性エネルギー蓄積を変更することができる.
The titin folding reaction can be biochemically tuned
タイチンは筋肉の純粋な機械的蛋白質でもなければ構造蛋白質でもない.それは筋肉における化学的かつ物理的ストレスの積分器でもあり,シグナル経路の一部であり,最終的には遺伝子発現に影響する.タイチンの弾性セグメントは翻訳後修飾の主要な標的であると示された.タイチンの非構造部位内の残基のリン酸化はタイチンの弾性においては穏やかな変化であると示された一方で (97,98,99), さらに劇的な変化は I-band の Ig ドメインの標的化により達成される.マウス心筋梗塞モデルにおいては虚血再灌流障害による酸化ストレスはサルコメア蛋白質の S-グルタチオニル化を招き,それには非常に巨大な分子量の蛋白質が含まれ,タイチンであると主張されている (100). タイチン I-band の Ig ドメインは不釣り合いに高いシステイン残基を含有している.しかし,これら大半のシステイン側鎖は謎めいている.それらは Ig 折り畳みの疎水性コアに埋め込まれており,溶媒にアクセスできないことを意味している.それらは Ig ドメインが伸展し始めない限り, S-グルタチオニル化できない. Alegre-Cebollada とその仲間が示したところでは,単分子 force-clamp AFM を用いて,単分子タイチン I91 ドメインの S-グルタチオニル化が力によりアンフォールディングした (78). ある重大な発見があった. S-グルタチオニル化が I91 ドメインの疎水性コア内部に好ましくない立体構造を作り出し,それがフォールディングを阻害したのである.ゆえにそれは酸化型グルタチオンは単一心筋内の受動的張力を大幅に減少させることができると証明することになったが,心筋細胞が伸展して Ig ドメインを動員し,アンフォールディング状態になった後に限られる.リン酸化のように, S-グルタチオニル化は可逆的であり,筋組織内部で酵素的な切断や変性を通じてグルタチオンを減少させることさえする.しかし,リン酸化はタイチン分子の硬度または Kuhn 長において小さな変化しかもたらさない一方で, Ig 折り畳みの S-グルタチオニル化や他の非特異的酸化はフォールディング反応に大規模な閉塞をもたらすことができる.仮に全タイチンのフォールディングが阻害されたら,その弾力特性は Figure 4B に示した青の点線のようにふるまうだろう.受動的張力の喪失は他のサルコメア蛋白質により感知されるところとなり,細胞シグナリングに変換される結果となると予想される.他の主要なタイチンの酸化的修飾は,分子内のジスルフィド結合である.そのジスルフィド結合は,機械的安定性やアンフォールディング率への大幅な影響なしに, Ig ドメインが折りたたまれる箇所での,大幅な力の増加を可能とする (80). フォールディング確率シフトの結果,フォールディング反応が収縮時により高い力を発生させる結果となる (Figure 4B, 金色曲線). 酸化ストレスに加えて,アシドーシスが筋肉の恒常性に対する一般的な摂動であり,蛋白質フォールディングに直接的な結果をもたらす (101). pH の変化は多くの側鎖のプロトン化を変化させ,それは順番に静電気力および疎水力を変化させ,蛋白質フォールディングを駆動し,機械的安定性を提供する.アシドーシスはフォールディングを阻害するかミスフォールディングを誘導し,それはフォールディング収縮の拡大を減弱させて筋弾性の変化をもたらす (102). 新たな視点は次のようなものでもある.つまり,筋特異的シャペロンは物理的伸展に対してタイチンの I-band セグメントに動員される可能性があり (103), ある種の筋疾患においてレベルがひどく変更される分子シャペロンがある (104) ということである.これらのシャペロンのいくつか,例えば αB-クリスタリンは,折りたたまれた Ig ドメインに直接結合することが可能であり,それらをアンフォールディングに対してより抵抗性にする (Figure 4B, 赤曲線) (103,105). 単分子原子間力分光法は最近分子シャペロンの可能性を明らかにし始めており,凝集を防ぐだけでなく,ある種の蛋白質のフォールディング反応を加速しているとされる (Haldar, 未公開データ). フォールディング力学における増加はタイチンフォールディング反応からの出力増加と同義であり,ゆえに 3 フィラメントモデルに従って筋収縮に直接影響を与えるだろう.
A rechargeable gravitational battery
ここで筋収縮の視点を,安静時の単独モーター活性から,伸展力に抵抗しながら機械的に伸展された蛋白質が折りたたまれて短縮する可能性に移そう.食物摂取を通じた太陽光から ATP へのエネルギー変換と動物細胞の代謝機構とは,筋収縮時に利用される化学エネルギーの源である.フォールディングした蛋白質により提供されるエネルギーとはなんだろう?短距離の疎水性崩壊と組み合わされたエントロピー反動が蛋白質折りたたみの力を供与すると我々は知っている.だが,蛋白質がフォールディングの仕事をなすことは明らかだが,その前に機械的に伸展されていなければならない.多くの折りたたみ可能な Ig ドメインへのタイチンポリペプチドの分節化はゆえに,ただの弾性収縮力のみを有する単純なばねから Ig ドメインを分化させる.この新しい機構は魅力的である.というのも,化学エネルギーに加えて,筋肉は他の基本的な力を利用するからである.重力と慣性は,動物がランニングなどの運動中に筋肉のタイチンを伸展しアンフォールディングさせ,エントロピー力と疎水力は負荷減少中に伸展した蛋白質の崩壊とフォールディングを駆動する (34,106). 筋サルコメアはゆえに,タイチン Ig ドメインが伸展した形態で環境からの機械的エネルギーを吸収し蓄積するのに最適に設計されており,この機械的エネルギーを化学的に活性化したミオシン II 型モーターによりトリガーされた時に放出する (Figure 3B). ミオシンモーターが前進できないほど高い負荷がかかっている時 (6 pN) でもタイチンは容易に折りたたまれる.ゆえに,タイチンフォールディングは失速したミオシンモーターを解放し,それらが前進する速度を設定し,効率的な筋収縮を許す.
筋収縮におけるタイチンのこの活動的な役割は,バッテリーのアナロジーで理解できる (Figure 5A 参照). 伸展フェーズでの Ig ドメインのアンフォールディングは外力が 10 pN を超える時に「バッテリー」に収縮エネルギーを充電する.モーターが参加した時に外力が弱まるにつれて Ig ドメイン再フォールディングは蓄積したエネルギーを放出し,引き続いて筋収縮がミオシンモーターによりなされる仕事と共に起きる.それゆえ,タイチンとミオシンは伸展フェーズでバッテリーが充電される (Figure 5A, 黒線) ハイブリッドシステムの二つの要素であり, ATP 化学エネルギーにより駆動されるミオシン頭部が参加した後にのみ,この蓄積エネルギーは放出される (Figure 5A, 赤線). にもかかわらず,収縮エネルギーが蓄積され放出される確率はむしろ異なり, Figure 5A に示すように異なる斜線に反映される. 10 pN でのタイチンアンフォールディングは 1.5 × 10-4 までの確率で発生し (65), ゆえにタイチンは少ない力で収縮エネルギーを蓄積し,その力は 0.02 zW と推定されている(10 pN (140 zJ) での Ig ドメインアンフォールディングの仕事を観察すると 2 倍の確率である). しかし,再フォールディングは更に高い確率で発生し,ドメインフォールディングは 7 秒間で 6 pN であり,タイチン Ig ドメイン再フォールディングで放出されるその力は約 100 倍も大きく, 6.5 zW にも達すると推定されている.その力の関係は異なるドメインによるものであろうが,生体内での力の放出はより複雑であるに違いない.動物の運動において筋肉がどうやって動作するかを極端に単純化すると,ヒラメ筋のような脚の筋肉はその静止状態から伸展位置に移動し,高いエネルギーをタイチンに変換する (Figure 5A, 下部パネル 1 から 2). 後に,分子モーターと ATP 加水分解が参加した後にタイチンに変換される力は減少し,タイチンから機械的仕事が放出されて筋肉が収縮する (Figure 5A, 下部パネル 2 から 3). タイチンドメインのフォールディング・アンフォールディング上のエネルギー変換の意味からは,上記で提案した蓄積・放出サイクルは,タイチン分子の自由エネルギーを背景にした融合プロセスとして描くことができる (Figure 5B). 12 pN での(緑色背景)蓄積フェーズ中そのエネルギー背景は伸展状態に傾いており(大伸展),そのためタイチンドメインが伸展し,順次自由エネルギー障壁を越えるのを願っている.外力が 6 pN にまで減少すると(赤色背景),その分子は再フォールディングにより短縮し,そのドメインは反対方向に障壁を願い,ゆえに収縮エネルギーを放出する.
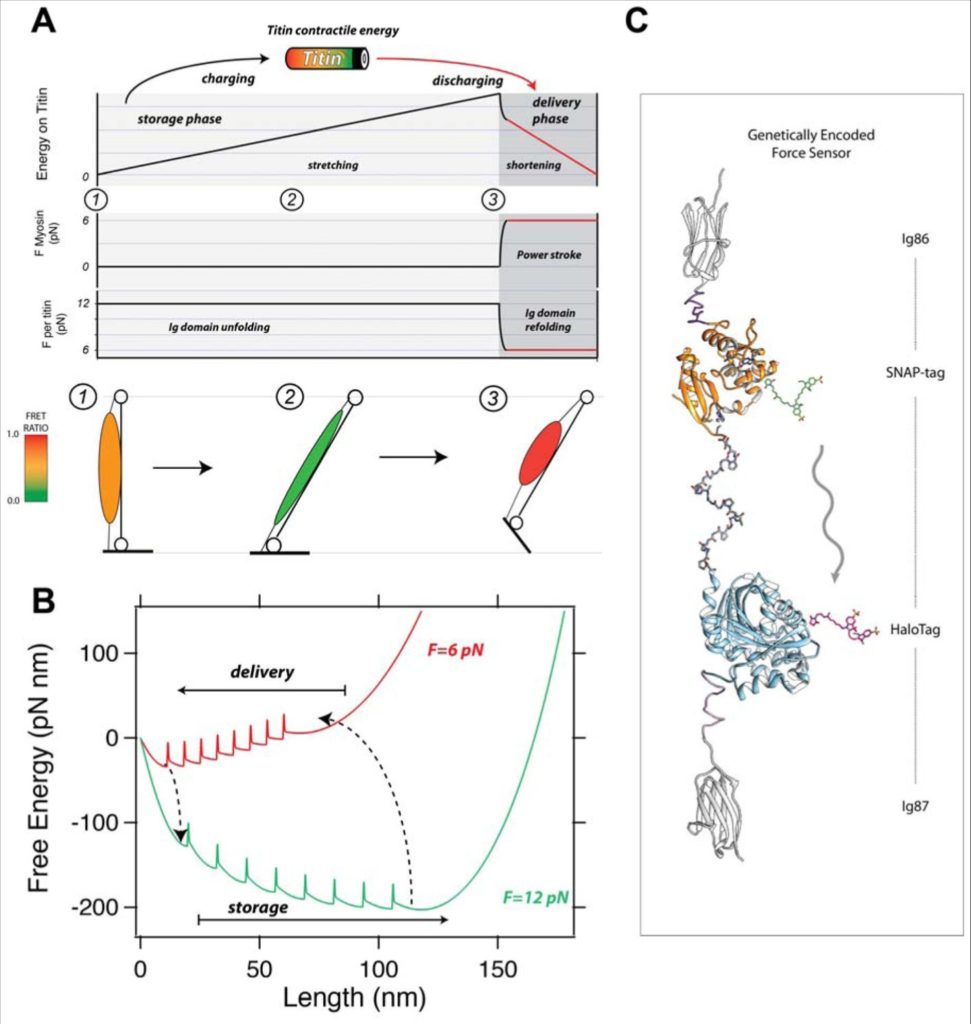
(A) 筋収縮におけるタイチンフォールディング・アンフォールディングの役割(パネル上部). タイチンドメインは「バッテリー」として機能し,収縮エネルギーをアンフォールディングにより蓄え,フォールディングの短縮時に供給する.筋肉が伸展する時,機械的張力が増加し,タイチンドメインがアンフォールドする(蓄積フェーズ (1)-(2)). 筋肉が収縮すると、ミオシン頭部が従事してタイチンにかかる力が 6 pN に低下し, Ig ドメインが再フォールディングして短縮し,収縮エネルギーが供給されて筋収縮を補助する (3)(下部パネル).運動中の動物の筋肉の伸張-収縮のための簡略化されたモデル,フォールディングおよび再フォールディング時の三相のタイチンを示す.(B) タイチン補助の筋収縮の自由エネルギー景観解釈.伸展フェーズにおいてはその景観は増加する力によりアンフォールド状態(より長い長さ)に傾き (12 pN, 青色), タイチンは収縮エネルギーをそのドメインのアンフォールディングにより蓄積する.ミオシンモーターが参加する時,タイチンにかかる負荷は 6 pN に減少するため,その景観は折りたたまれた状態に有利となり,自由エネルギー障壁を乗り越えてドメインを折りたたむことが可能となり,分子全長を短縮して収縮の仕事を提供することが可能となる.(C)この仮説を試験するには,遺伝的にコード化された力学センサーをタイチン配列に組み込むことが必要である.このような設計の一つは,エキソン I86 と I87 との間に導入された HaloTag および SNAP タグ共有標識技術を利用して提供される.仮説的な緑色供与体蛍光団および赤色アクセプター蛍光団を用いて,パネル (A) に示される歩みを通して,筋肉の FRET 比は最大から最小に変化する.これは生体内でのタイチン 1 分子あたりにかかる力を直接読み出すことを可能とする.
我々の現在の観察の限界は磁気ピンセットで探索した 8 回繰り返し Ig ドメインの 6 pN での遅いフォールディング率である (Figure 1B). 実際,単分子の観察を生体組織に外挿する時には我々は慎重でなくてはならない.これらの観察が複雑なサルコメアの環境においてどのように再生されるのか,我々はまだ完全に理解しているわけではないからである.しかし,いくつかの単純な観察は保証されている.伸展したドメインに加えて,野生型のタイチンは巨大な非構造セグメントを有しており,例えば PEVK や N2B などだが,これらはタイチンがポリマー拡散の限界速度でサルコメア内のいかなる外力にも平衡することを許しており,その速度はどんな最速の筋収縮速度よりも速い (73). 実際,伸展長からの受動的サルコメア短縮は最初は急速であり (107,108), 最近の生体内筋顕微鏡では,サルコメアはタイチンが数 pN の構成的な力の下での長さにおいて動作するようである.これのさらに示唆するところは,タイチンは弛緩するのではないということである (90,92,93). より重要なことは,短い,単分子実験でよく用いられる 8 回繰り返し Ig の操作された蛋白質とは対照的に,野生型タイチンの拡張可能なセグメントは 50-100 のアイソフォーム依存性 Ig ドメインを含有しており,外力減少に伴い初期フォールディング率が急激に加速する.生体内で観測される相対的に短いサルコメア長の変化を超え,例えば心筋組織におけるサルコメア半分あたり 50 nm までなど,その初期のフォールディング率はタイチンフォールディングの生理学的に関連する出力のより良い計測を可能にしてくれる.これらの効果を垣間見ることは,試験管内でのタイチン力学を計測可能とするものであり,タイチンフォールディング事象が 100 ミリ秒以下の時間軸で発生することを示している (10). 最後に,サルコメアはタイチン特異的シャペロンで混雑した環境であり,それが我々の測定よりもさらにフォールディング率を増加させているに違いない (102,103). これらの効果の全てが, Figure 1B に示した実験では欠けているが,サルコメア内部でのタイチンフォールディングの出力を強化するだろう.
Synchronization of titin folding with actomyosin contraction
タイチンのための我々の仮説は筋収縮状態を強化するものである.つまり,タイチンとミオシンは協力して働く.それはタイチンフォールディングが架橋の参与により引き起こされる外力の減少に依存しているためである.このメカニズムの示唆するところは,太いフィラメントに付着している 6 つのタイチン分子は同時に折りたたまれるだろうということである.単分子原子間力分光法の試験は平行なポリ蛋白質のスケーリングの法則を証明した.蛋白質ドメインが平行に伸展される時,それらは同時に破裂し,力は二倍となり,永続長は半分となる (109,110). それゆえ,我々の示唆するところは,6 本のタイチン Ig ドメインも同時に折りたたまれ,それは太いフィラメント周囲の平行に配列されていることによる,というものである. 6 本の平行なタイチン Ig ドメインの最初の一本が確率的に折りたたまれる時,他の 5 本にかかる力は即座に減少する.フォールディングやアンフォールディング率は外力に指数関数的に依存し,外力のほんの少しの減少でさえ,他の平行なドメインのフォールディングを加速するだろう.ゆえに,タイチン Ig フォールディングからの出力は高度に協調的である.さらに,アクチン-ミオシン収縮が同期しているのにタイチンはそうではない,と言うのは誤解を招く (111). 次の点は一般に受け入れられている.つまり,筋肉短縮に駆動されるミオシンは “サイクリック非同期 ATP 駆動形アクチン-ミオシン収縮” により発生すると (112). 架橋形成はミオシン頭部とアクチンフィラメントの拡散遭遇に依存しており,確率的プロセスであると定義される.加えて, ATP 加水分解により放出されるエネルギーは kT より大きなオーダーの大きさに過ぎないため,その後のミオシンの構造変化は,少なくとも部分的には,ブラウン運動に依存する (113). この視点から,ミオシンモーターのサイクリングとタイチン Ig ドメインのフォールディングとは共に確率的であるが,我々のモデルでは,筋肉の化学的活性化において同時に発生する.
Testable predictions and the techniques to examine them
筋肉は複雑なシステムであり,階層構造に配列された蛋白質のいくつかの相互作用を含んでいる.分子レベルからこれらの各蛋白質の特定の役割を定義することは困難な事業である.しかし,タイチン分子のタンデムモジュール式配置は筋肉の受動的張力の役割に単に専念するだけではなく,実際にこれらのドメインが外力の範囲内で伸展し再度折りたたまれて動作することは,今や明らかになったように見える.筋肉の短縮時にあたり,これは収縮の仕事を発生させるタイチンの活動機能を伴う.しかし,筋収縮におけるタイチンの精密な役割および,いかにしてドメインフォールディングの出力がミオシンモーターの出力と相互作用するかの詳細なビジョンを提供するために,多くの実験の仕事が残されている.全体を通じて議論された仮説は検証可能な予測の鍵を作る.つまり,筋肉の活性化はミオシンモーターの参加へと繋がり,タイチンへの負荷を減少させて,以前に伸展状態に動員された Ig ドメインを折りたたむトリガーとなる (10). 我々は自分自身に問わなければならない.タイチンのこのエネルギッシュな貢献はいかにして分離したのか?また,現在の生物物理学的ツールは十分なのか?と.
原子間力分光学者たちは,全組織レベルでの単分子技術の精度をもたらしてくれるツールを持つことを心から望んでいる.ある程度,これは一般にはコード化された力学センサーと共に認識されてきており,培養細胞の力を支える細胞骨格成分内部で発現している (114). これらの力学センサーは二種の蛍光蛋白質を用い,非放射性移動を通じてエネルギーを変換することができ (FRET), 短い非構造化または構造化ペプチドに分離している.これらの一般にコード化された力学センサーはスペクトリン蛋白 (115), ビンキュリンおよびタリン (116,117), そして核ネスプリン (118) に組み込まれ,これらは全て細胞内ストレス線維からの張力の材料である.力学センサーを有する蛋白質にかかる張力が増加するにつれ, FRET ドナーおよびアクセプターは分離して FRET 率が低下する.これらの力学センサーは FRET 率を計測することでキャリブレーションされ,それは 0-10 pN の範囲を超える単分子原子間力分光法を用いて適用された外力の下での FRET 率である (119). タイチン I-band は技術者にとって Figure 5 に描かれたように力学センサーの理想的な部位になりうる.細胞培養実験と異なり,制御されていない方法で細胞接着後の細胞骨格張力が増加するところでは,タイチンにかかるその張力は筋線維牽引装置により絶妙に調節される可能性がある (9,108,120). タイチンフィラメントのサルコメア内部での統一された配置は,発色団が高度に整列することを確保しており,無傷の筋肉内の蛍光読み取りの優れたシグナルノイズ比を提供してくれる.最も重要なことだが,タイチンにかかる外力はバス内のカルシウム濃度上昇により調節される可能性があり,その結果架橋形成がタイチンから力を短絡させる結果となることを我々は示唆した.筋収縮における我々の 3 フィラメントモデルに従うと,生理的ストレッチの下での筋肉内の架橋形成は各タイチンにかかる力を 10 pN 以下に減少させ,そこでタイチンフォールディングがピーク出力を放出する.一般的にコード化された力学センサーがタイチンごとの力を決定することを可能とするだけではなく,タイチンフォールディング収縮でのサルコメア長もまたトリガーされる.各筋群は異なる長さのタイチンアイソフォームを有している.例えば異なる 40 セットのウサギの筋肉のように (87). 我々の示唆するところでは,この長さは次のようにプログラムされている.つまり,タイチンフォールディング収縮は,特定の筋肉群の最適な仕事をするサルコメア長の中で発生すると.そのようなツールは筋肉の動作の研究にとって,生体内や試験管内いずれにおいても非常に貴重であり,タイチンおよびタイチンにかかる負荷を減少させるのに必要な架橋の数により経験する生理学的な力をより良く理解するための分野への移動の助けとなるだろう.これらの二つの力は,タイチンのフォールディング速度と併せて,筋収縮時のタイチンとミオシンの相対的な出力を決定するのに重要である.
そのような生体内ツールの発展への最初の段階は達成されている. I-band 遠位部分への HaloTag 酵素のノックインを有するモデルマウスはタイチンの生物物理学的特性のための多目的ツールとして奉仕した (121). HaloTag 酵素の使用は,ガラス製カバースリップに単分子を牽引する実験のための共有結合アンカー蛋白質として,我々のラボで開発されたものであり (41,49), 蛍光イメージングの広範囲の取得を可能とした (122,123). HaloTag はクロロアルカン基質を認識して共有結合し,様々な有機発色団と付着して優れたイメージング特性を持つ.抗体と違って,クロロアルカンリガンドは非常に小さく,容易にサルコメアに浸透して高濃度の標識に達することができる. SNAP-Tag, CLIP-Tag や TMP-tag (122) などの直交標識部位を HaloTag に続けて配置することで,上述したセンサーに近似した一般的にコード化されたセンサーが達成される可能性がある (Figure 5C). これは先述した有機染料を用いることができる点で一般的にコード化された力学センサーに対して優位性があり,蛍光蛋白質のセンサーより優れた特性を持つ.使用した蛍光発色団のセットは手元の実験でも適応する可能性があり,単一筋線維伸展実験や生体内イメージングにも適応する可能性がある. HaloTag に加えて,現在のマウスモデルは TEV (ウイルス)プロテアーゼ部位をも有している.これまでのタイチン分子を切断しようとする試み (107,124,125) と違って,これはタイチン分子の半分(ノックインのヘテロ接合体マウス)か,全タイチン分子(ホモ接合体)を高度に特異的に切断することが可能であり,それにより筋弾性におけるタイチンの役割と力の発生をよりよく理解することができる.
筋収縮の複雑さをより深く探求することは古典的な筋収縮実験の再評価を要する.それには筋肉の受動的および能動的メカニズムの両者においてタイチンが主要な役割を果たすことを念頭に置く必要がある.ミオシン頚部の追従における下方傾向を鑑みるに (89,126,127,128), 収縮するサルコメアの主要なエネルギー蓄積源は再考されなくてはならない.おそらく一般にコード化された力学センサーは, Huxley と Simmons の古典的なクイックリリース実験 (9) のバージョンを再考することで,タイチンが主要なエネルギー蓄積源であることを証明することになるだろう.そして再構築されたアクチンミオシン複合体の単分子実験が改善しつつあり (26,28,29), さらにミオシンによる力の発生メカニズムへの洞察をもたらしつつあるにもかかわらず,無傷のサルコメア内部で単一のミオシンモーターの速度やストロークサイズを直接観察した実験はまだ存在しない.この観点からは,そのミオシンモーターの分野はタイチンより後の段階である.さらに,熱力学エンジンのように筋肉を扱うことは, Fenn や Wilkie その他の研究者らが熱電堆と熱量計を用いて筋肉エンタルピーを計測した結果,ミオシンモーター単独に熱産生を割り当ててしまったように,問題を過度に単純化してしまう (27,129). 単純な粘弾性ポリマーは力の減少に乗じて巨大なエントロピー増大を経て,周辺環境から熱を吸収する.折りたたまれた Ig ドメインは単純な粘弾性ポリマーではない.ミオシンモーターが参加してタイチンにかかる外力が減少する時,まずエントロピー増加が起きてからタイチンが熱を吸収するに違いない.しかし,モルテン・グロビュール遷移の崩壊は,それ自体がフォールディング収縮の主要な一部なのだが,高度にコンパクトな形態を取り,低いエントロピーでの高次蛋白質に折りたたまれる結果となる.ゆえに,蛋白質フォールディングはエントロピーの純損失となる可能性があり,周辺のバスへの熱喪失の結果となる. Fenn らの結果は今や再考される必要があり,我々はタイチン蛋白質フォールディングの熱力学が粘弾性ポリマーほど単純ではないことを知っている.
結論として,筋収縮における我々の視点は,安静単独のモーター活性から,伸展力に抗して折りたたまれる機械的に展開した蛋白質の可能性に焦点を移した.タイチンのこのユニークな素材特性は,その折りたたみ可能な Ig ドメインにより与えられ, Hookean ばねとも非構造ポリマーとも異なり,唯一,弾性収縮を経験する受動的エラストマーであることだろう.単分子データは明らかに示しているが, 6 pN 以上では,ミオシンモーターは失速して前進できず,機械的仕事をなすことができない.それに応じて,タイチン Ig ドメインはその外力の範囲で容易に再折りたたみ,収縮の仕事を提供して失速したミオシンモーターを解放し,効果的な筋収縮を許す.ゆえに,この新しい根拠の示すところは,第三のフィラメントとして認識されたのと同様に,筋収縮においてアクチンとミオシンと共に働く不可分のパートナーとして,タイチンをサルコメアの活性要素として考慮する必要がある.
References
1. Maruyama K, Matsubara S, Natori R, Nonomura Y, Kimura S, et al. Connectin, an Elastic Protein of Muscle Characterization and Function. J Biochem (Tokyo). 1977; 82(2):317–37. [PubMed: 914784]
2. Polissar MJ. Physical chemistry of contractile process in muscle. I. A physicochemical model of contractile mechanism. Am J Physiol. 1952; 168(3):766–81. [PubMed: 14914890]
3. Polissar MJ. Physical chemistry of contractile process in muscle. II. Analysis of other mechanochemical properties of muscle. Am J Physiol. 1952; 168(3):782–92. [PubMed: 14914891]
4. Polissar MJ. Physical chemistry of contractile process in muscle. III. Interpretation of thermal behavior of stimulated muscle. Am J Physiol. 1952; 168(3):793–804. [PubMed: 14914892]
5. Polissar MJ. Physical chemistry of contractile process in muscle. IV. Estimates of size of contractile unit. Am J Physiol. 1952; 168(3):805–11. [PubMed: 14914893]
6. Gordon AM, Huxley AF, Julian FJ. Tension development in highly stretched vertebrate muscle fibres. J Physiol. 1966; 184(1):143–69. [PubMed: 5921535]
7. Gordon AM, Huxley AF, Julian FJ. The variation in isometric tension with sarcomere length in vertebrate muscle fibres. J Physiol. 1966; 184(1):170–92. [PubMed: 5921536]
8. Huxley AF, Niedergerke R. Structural Changes in Muscle During Contraction: Interference Microscopy of Living Muscle Fibres. Nature. 1954; 173(4412):971–73. [PubMed: 13165697]
9. Huxley AF, Simmons RM. Proposed Mechanism of Force Generation in Striated Muscle. Nature. 1971; 233(5321):533–38. [PubMed: 4939977]
10. Rivas-Pardo JA, Eckels EC, Popa I, Kosuri P, Linke WA, Fernández JM. Work Done by Titin Protein Folding Assists Muscle Contraction. Cell Rep. 2016; 14(6):1339–47. [PubMed: 26854230]
11. Huxley HE. Structural Arrangements and the Contraction Mechanism in Striated Muscle. Proc R Soc Lond B Biol Sci. 1964; 160(981):442–48. [PubMed: 14214772]
12. Maruyama K, Natori R, Nonomura Y. New elastic protein from muscle. Nature. 1976; 262(5563): 58–60. [PubMed: 934326]
13. Maruyama K, Kimura S, Ohashi K, Kuwano Y. Connectin, an Elastic Protein of Muscle. Identification of “Titin” with Connectin. J Biochem (Tokyo). 1981; 89(3):701–9. [PubMed: 7287636]
14. Stull JT. Myosin Minireview Series. J Biol Chem. 1996; 271(27):15849–15849. [PubMed: 8663555]
15. Bang M-L, Centner T, Fornoff F, Geach AJ, Gotthardt M, et al. The Complete Gene Sequence of Titin, Expression of an Unusual ≈700-kDa Titin Isoform, and Its Interaction With Obscurin Identify a Novel Z-Line to I-Band Linking System. Circ Res. 2001; 89(11):1065–72. [PubMed: 11717165]
16. Burkholder TJ, Lieber RL. Sarcomere length operating range of vertebrate muscles during movement. J Exp Biol. 2001; 204(9):1529–36. [PubMed: 11296141]
17. Millman BM. The Filament Lattice of Striated Muscle. Physiol Rev. 1998; 78(2):359–91. [PubMed: 9562033]
18. Cazorla O, Freiburg A, Helmes M, Centner T, McNabb M, et al. Differential Expression of Cardiac Titin Isoforms and Modulation of Cellular Stiffness. Circ Res. 2000; 86(1):59–67. [PubMed: 10625306]
19. Bogomolovas J, Gasch A, Simkovic F, Rigden DJ, Labeit S, Mayans O. Titin kinase is an inactive pseudokinase scaffold that supports MuRF1 recruitment to the sarcomeric M-line. Open Biol. 2014; 4(5):140041. [PubMed: 24850911]
20. Lange S, Xiang F, Yakovenko A, Vihola A, Hackman P, et al. The Kinase Domain of Titin Controls Muscle Gene Expression and Protein Turnover. Science. 2005; 308(5728):1599–1603. [PubMed: 15802564]
21. Linke WA, Hamdani N. Gigantic Business Titin Properties and Function Through Thick and Thin. Circ Res. 2014; 114(6):1052–68. [PubMed: 24625729]
22. Hessel AL, Lindstedt SL, Nishikawa KC. Physiological Mechanisms of Eccentric Contraction and Its Applications: A Role for the Giant Titin Protein. Front Physiol. 2017; 8
23. Tskhovrebova L, Trinick J. Titin: properties and family relationships. Nat Rev Mol Cell Biol. 2003; 4(9):679–89. [PubMed: 14506471]
24. Linke WA, Krüger M. The Giant Protein Titin as an Integrator of Myocyte Signaling Pathways. Physiology. 2010; 25(3):186–98. [PubMed: 20551232]
25. Spudich JA. Hypertrophic and Dilated Cardiomyopathy: Four Decades of Basic Research on Muscle Lead to Potential Therapeutic Approaches to These Devastating Genetic Diseases. Biophys J. 2014; 106(6):1236–49. [PubMed: 24655499]
26. Finer JT, Simmons RM, Spudich JA. Single myosin molecule mechanics: piconewton forces and nanometre steps. Nature. 1994; 368(6467):113–19. [PubMed: 8139653]
27. Wilkie DR. Heat work and phosphorylcreatine break-down in muscle. J Physiol. 1968; 195(1):157– 83. [PubMed: 5639798]
28. Kaya M, Higuchi H. Nonlinear Elasticity and an 8-nm Working Stroke of Single Myosin Molecules in Myofilaments. Science. 2010; 329(5992):686–89. [PubMed: 20689017]
29. Kaya M, Tani Y, Washio T, Hisada T, Higuchi H. Coordinated force generation of skeletal myosins in myofilaments through motor coupling. Nat Commun. 2017; 8 ncomms16036.
30. Herzog W, Schappacher G, DuVall M, Leonard TR, Herzog JA. Residual Force Enhancement Following Eccentric Contractions: A New Mechanism Involving Titin. Physiology. 2016; 31(4): 300–312. [PubMed: 27252165]
31. Herzog W, Leonard TR. Force enhancement following stretching of skeletal muscle. J Exp Biol. 2002; 205(9):1275–83. [PubMed: 11948204]
32. Nishikawa KC, Monroy JA, Uyeno TE, Yeo SH, Pai DK, Lindstedt SL. Is titin a “winding filament”? A new twist on muscle contraction. Proc R Soc Lond B Biol Sci. 2012; 279(1730):981– 90.
33. Carrion-Vazquez M, Oberhauser AF, Fowler SB, Marszalek PE, Broedel SE, et al. Mechanical and chemical unfolding of a single protein: A comparison. Proc Natl Acad Sci. 1999; 96(7):3694–99. [PubMed: 10097099]
34. Fernandez JM, Li H. Force-Clamp Spectroscopy Monitors the Folding Trajectory of a Single Protein. Science. 2004; 303(5664):1674–78. [PubMed: 15017000]
35. Kellermayer MSZ, Smith SB, Granzier HL, Bustamante C. Folding-Unfolding Transitions in Single Titin Molecules Characterized with Laser Tweezers. Science. 1997; 276(5315):1112–16. [PubMed: 9148805]
36. Li H, Linke WA, Oberhauser AF, Carrion-Vazquez M, Kerkvliet JG, et al. Reverse engineering of the giant muscle protein titin. Nature. 2002; 418(6901):998–1002. [PubMed: 12198551]
37. Rief M, Gautel M, Oesterhelt F, Fernandez JM, Gaub HE. Reversible Unfolding of Individual Titin Immunoglobulin Domains by AFM. Science. 1997; 276(5315):1109–12. [PubMed: 9148804]
38. Schlierf M, Li H, Fernandez JM. The unfolding kinetics of ubiquitin captured with single-molecule force-clamp techniques. Proc Natl Acad Sci U S A. 2004; 101(19):7299–7304. [PubMed: 15123816]
39. Tskhovrebova L, Trinick J, Sleep JA, Simmons RM. Elasticity and unfolding of single molecules of the giant muscle protein titin. Nature. 1997; 387(6630):308–12. [PubMed: 9153398]
40. Popa I, Kosuri P, Alegre-Cebollada J, Garcia-Manyes S, Fernandez JM. Force dependency of biochemical reactions measured by single-molecule force-clamp spectroscopy. Nat Protoc. 2013; 8(7):1261–76. [PubMed: 23744288]
41. Popa I, Berkovich R, Alegre-Cebollada J, Badilla CL, Rivas-Pardo JA, et al. Nanomechanics of HaloTag Tethers. J Am Chem Soc. 2013; 135(34):12762–71. [PubMed: 23909704]
42. Chen H, Fu H, Zhu X, Cong P, Nakamura F, Yan J. Improved High-Force Magnetic Tweezers for Stretching and Refolding of Proteins and Short DNA. Biophys J. 2011; 100(2):517–23. [PubMed: 21244848]
43. Chen H, Yuan G, Winardhi RS, Yao M, Popa I, et al. Dynamics of Equilibrium Folding and Unfolding Transitions of Titin Immunoglobulin Domain under Constant Forces. J Am Chem Soc. 2015; 137(10):3540–46. [PubMed: 25726700]
44. Svoboda K, Schmidt CF, Schnapp BJ, Block SM. Direct observation of kinesin stepping by optical trapping interferometry. Nature. 1993; 365(6448):721–27. [PubMed: 8413650]
45. Martonfalvi Z, Bianco P, Linari M, Caremani M, Nagy A, et al. Low-force transitions in single titin molecules reflect a memory of contractile history. J Cell Sci. 2014; 127(4):858–70. [PubMed: 24357719]
46. Neuman KC, Nagy A. Single-molecule force spectroscopy: optical tweezers, magnetic tweezers and atomic force microscopy. Nat Methods. 2008; 5(6):491–505. [PubMed: 18511917]
47. Huhle A, Klaue D, Brutzer H, Daldrop P, Joo S, et al. Camera-based three-dimensional real-time particle tracking at kHz rates and Ångström accuracy. Nat Commun. 2015; 6
48. Dulin D, Cui TJ, Cnossen J, Docter MW, Lipfert J, Dekker NH. High Spatiotemporal-Resolution Magnetic Tweezers: Calibration and Applications for DNA Dynamics. Biophys J. 2015; 109(10): 2113–25. [PubMed: 26588570]
49. Popa I, Rivas-Pardo JA, Eckels EC, Echelman DJ, Badilla CL, et al. A HaloTag Anchored Ruler for Week-Long Studies of Protein Dynamics. J Am Chem Soc. 2016; 138(33):10546–53. [PubMed: 27409974]
50. Bustamante C, Marko JF, Siggia ED, Smith S. Entropic elasticity of lambda-phage DNA. Science. 1994; 265(5178):1599–1600. [PubMed: 8079175]
51. Rief M, Clausen-Schaumann H, Gaub HE. Sequence-dependent mechanics of single DNA molecules. Nat Struct Mol Biol. 1999; 6(4):346–49.
52. Rief M, Oesterhelt F, Heymann B, Gaub HE. Single Molecule Force Spectroscopy on Polysaccharides by Atomic Force Microscopy. Science. 1997; 275(5304):1295–97. [PubMed: 9036852]
53. Marszalek PE, Oberhauser AF, Pang Y-P, Fernandez JM. Polysaccharide elasticity governed by chair–boat transitions of the glucopyranose ring. Nature. 1998; 396(6712):661–64. [PubMed: 9872313]
54. Florin EL, Moy VT, Gaub HE. Adhesion forces between individual ligand-receptor pairs. Science. 1994; 264(5157):415–17. [PubMed: 8153628]
55. Radmacher M, Fritz M, Hansma HG, Hansma PK. Direct observation of enzyme activity with the atomic force microscope. Science. 1994; 265(5178):1577–79. [PubMed: 8079171]
56. Carrion-Vazquez M, Marszalek PE, Oberhauser AF, Fernandez JM. Atomic force microscopy captures length phenotypes in single proteins. Proc Natl Acad Sci. 1999; 96(20):11288–92. [PubMed: 10500169]
57. Valle-Orero J, Rivas-Pardo JA, Tapia-Rojo R, Popa I, Echelman DJ, et al. Mechanical Deformation Accelerates Protein Ageing. Angew Chem Int Ed. n/a-n/a.
58. Oberhauser AF, Hansma PK, Carrion-Vazquez M, Fernandez JM. Stepwise unfolding of titin under force-clamp atomic force microscopy. Proc Natl Acad Sci. 2001; 98(2):468–72. [PubMed: 11149943]
59. Wiita AP, Ainavarapu SRK, Huang HH, Fernandez JM. Force-dependent chemical kinetics of disulfide bond reduction observed with single-molecule techniques. Proc Natl Acad Sci. 2006; 103(19):7222–27. [PubMed: 16645035]
60. Wiita AP, Perez-Jimenez R, Walther KA, Gräter F, Berne BJ, et al. Probing the chemistry of thioredoxin catalysis with force. Nature. 2007; 450(7166):124–27. [PubMed: 17972886]
61. Ainavarapu SRK, Brujić J, Huang HH, Wiita AP, Lu H, et al. Contour Length and Refolding Rate of a Small Protein Controlled by Engineered Disulfide Bonds. Biophys J. 2007; 92(1):225–33. [PubMed: 17028145]
62. Sotomayor M, Schulten K. Single-Molecule Experiments in Vitro and in Silico. Science. 2007; 316(5828):1144–48. [PubMed: 17525328]
63. Flory, PJ. Principles of Polymer Chemistry. Cornell University Press; 1953. p. 696
64. Stacklies W, Vega MC, Wilmanns M, Gräter F. Mechanical Network in Titin Immunoglobulin from Force Distribution Analysis. PLOS Comput Biol. 2009; 5(3):e1000306. [PubMed: 19282960]
65. Yuan G, Le S, Yao M, Qian H, Zhou X, et al. Elasticity of the Transition State Leading to an Unexpected Mechanical Stabilization of Titin Immunoglobulin Domains. Angew Chem Int Ed. 2017 n/a-n/a.
66. Berkovich R, Garcia-Manyes S, Klafter J, Urbakh M, Fernández JM. Hopping around an entropic barrier created by force. Biochem Biophys Res Commun. 2010; 403(1):133–37. [PubMed: 21050839]
67. Garcia-Manyes S, Dougan L, Badilla CL, Brujić J, Fernández JM. Direct observation of an ensemble of stable collapsed states in the mechanical folding of ubiquitin. Proc Natl Acad Sci. 2009; 106(26):10534–39. [PubMed: 19541635]
68. Redfield C, Smith RAG, Dobson CM. Structural characterization of a highly–ordered “molten globule” at low pH. Nat Struct Mol Biol. 1994; 1(1):23–29.
69. Mártonfalvi Z, Bianco P, Naftz K, Ferenczy GG, Kellermayer M. Force generation by titin folding. Protein Sci. 2017 n/a-n/a.
70. Eckels EC, Rivas-Pardo JA, Valle-Orero J, Popa I, Fernandez JM. The Science of Stretching: Mechanical Anisotropy in Titin Ig Domains. Biophys J. 2016; 110(3):393a.
71. Li H, Wang H-C, Cao Y, Sharma D, Wang M. Configurational Entropy Modulates the Mechanical Stability of Protein GB1. J Mol Biol. 2008; 379(4):871–80. [PubMed: 18472109]
72. Li H, Carrion-Vazquez M, Oberhauser AF, Marszalek PE, Fernandez JM. Point mutations alter the mechanical stability of immunoglobulin modules. Nat Struct Mol Biol. 2000; 7(12):1117–20.
73. Berkovich R, Hermans RI, Popa I, Stirnemann G, Garcia-Manyes S, et al. Rate limit of protein elastic response is tether dependent. Proc Natl Acad Sci. 2012; 109(36):14416–21. [PubMed: 22895787]
74. Berkovich R, Garcia-Manyes S, Urbakh M, Klafter J, Fernandez JM. Collapse Dynamics of Single Proteins Extended by Force. Biophys J. 2010; 98(11):2692–2701. [PubMed: 20513414]
75. Neupane K, Woodside MT. Quantifying Instrumental Artifacts in Folding Kinetics Measured by Single-Molecule Force Spectroscopy. Biophys J. 2016; 111(2):283–86. [PubMed: 27369870]
76. Brenner H. The slow motion of a sphere through a viscous fluid towards a plane surface. Chem Eng Sci. 1961; 16(3–4):242–51.
77. Valle-Orero J, Eckels EC, Stirnemann G, Popa I, Berkovich R, Fernandez JM. The elastic free energy of a tandem modular protein under force. Biochem Biophys Res Commun. 2015; 460(2): 434–38. [PubMed: 25796331]
78. Alegre-Cebollada J, Kosuri P, Giganti D, Eckels E, Rivas-Pardo JA, et al. S-Glutathionylation of Cryptic Cysteines Enhances Titin Elasticity by Blocking Protein Folding. Cell. 2014; 156(6): 1235–46. [PubMed: 24630725]
79. Li H, Fernandez JM. Mechanical Design of the First Proximal Ig Domain of Human Cardiac Titin Revealed by Single Molecule Force Spectroscopy. J Mol Biol. 2003; 334(1):75–86. [PubMed: 14596801]
80. Pardo JAR, Eckels E, Valle-Orero J, Fernadez JM. Disulfide Bonding in the Contractile Work of Titin. Biophys J. 2017; 112(3):456a.
81. Hill AV. The Heat of Shortening and the Dynamic Constants of Muscle. Proc R Soc Lond B Biol Sci. 1938; 126(843):136–95.
82. Debold EP, Patlak JB, Warshaw DM. Slip Sliding Away: Load-Dependence of Velocity Generated by Skeletal Muscle Myosin Molecules in the Laser Trap. Biophys J. 2005; 89(5):L34–36. [PubMed: 16169988]
83. Visscher K, Schnitzer MJ, Block SM. Single kinesin molecules studied with a molecular force clamp. Nature. 1999; 400(6740):184–89. [PubMed: 10408448]
84. Clemen AE-M, Vilfan M, Jaud J, Zhang J, Bärmann M, Rief M. Force-Dependent Stepping Kinetics of Myosin-V. Biophys J. 2005; 88(6):4402–10. [PubMed: 15764664]
85. Gennerich A, Carter AP, Reck-Peterson SL, Vale RD. Force-Induced Bidirectional Stepping of Cytoplasmic Dynein. Cell. 2007; 131(5):952–65. [PubMed: 18045537]
86. Linke WA, Stockmeier MR, Ivemeyer M, Hosser H, Mundel P. Characterizing titin’s I-band Ig domain region as an entropic spring. J Cell Sci. 1998; 111(11):1567–74. [PubMed: 9580564]
87. Prado LG, Makarenko I, Andresen C, Krüger M, Opitz CA, Linke WA. Isoform Diversity of Giant Proteins in Relation to Passive and Active Contractile Properties of Rabbit Skeletal Muscles. J Gen Physiol. 2005; 126(5):461–80. [PubMed: 16230467]
88. Campbell KS. Dynamic coupling of regulated binding sites and cycling myosin heads in striated muscle. J Gen Physiol. 2014; 143(3):387–99. [PubMed: 24516189]
89. Piazzesi G, Reconditi M, Linari M, Lucii L, Bianco P, et al. Skeletal Muscle Performance Determined by Modulation of Number of Myosin Motors Rather Than Motor Force or Stroke Size. Cell. 2007; 131(4):784–95. [PubMed: 18022371]
90. Llewellyn ME, Barretto RPJ, Delp SL, Schnitzer MJ. Minimally invasive high-speed imaging of sarcomere contractile dynamics in mice and humans. Nature. 2008; 454(7205):784–88. [PubMed: 18600262]
91. Rassier DE, MacIntosh BR, Herzog W. Length dependence of active force production in skeletal muscle. J Appl Physiol. 1999; 86(5):1445–1457. [PubMed: 10233103]
92. Cromie MJ, Sanchez GN, Schnitzer MJ, Delp SL. Sarcomere lengths in human extensor carpi radialis brevis measured by microendoscopy. Muscle Nerve. 2013; 48(2):286–92. [PubMed: 23813625]
93. Sanchez GN, Sinha S, Liske H, Chen X, Nguyen V, et al. In Vivo Imaging of Human Sarcomere Twitch Dynamics in Individual Motor Units. Neuron. 2015; 88(6):1109–20. [PubMed: 26687220]
94. Gittes F, Mickey B, Nettleton J, Howard J. Flexural rigidity of microtubules and actin filaments measured from thermal fluctuations in shape. J Cell Biol. 1993; 120(4):923–34. [PubMed: 8432732]
95. Fudge DS, Gardner KH, Forsyth VT, Riekel C, Gosline JM. The Mechanical Properties of Hydrated Intermediate Filaments: Insights from Hagfish Slime Threads. Biophys J. 2003; 85(3): 2015–27. [PubMed: 12944314]
96. Lovelady HH, Shashidhara S, Matthews WG. Solvent specific persistence length of molecular type I collagen. Biopolymers. 2014; 101(4):329–35. [PubMed: 23907817]
97. Hidalgo C, Hudson B, Bogomolovas J, Zhu Y, Anderson B, et al. PKC Phosphorylation of Titin’s PEVK Element A Novel and Conserved Pathway for Modulating Myocardial Stiffness. Circ Res. 2009; 105(7):631–38. [PubMed: 19679839]
98. Kötter S, Gout L, Frieling-Salewsky MV, Müller AE, Helling S, et al. Differential changes in titin domain phosphorylation increase myofilament stiffness in failing human hearts. Cardiovasc Res. 2013; 99(4):648–56. [PubMed: 23764881]
99. Perkin J, Slater R, Del Favero G, Lanzicher T, Hidalgo C, et al. Phosphorylating Titin’s Cardiac N2B Element by ERK2 or CaMKIIδ Lowers the Single Molecule and Cardiac Muscle Force. Biophys J. 2015; 109(12):2592–2601. [PubMed: 26682816]
100. Avner BS, Shioura KM, Scruggs SB, Grachoff M, Geenen DL, et al. Myocardial infarction in mice alters sarcomeric function via post-translational protein modification. Mol Cell Biochem. 2012; 363(1–2):203–15. [PubMed: 22160857]
101. Li Y, Linke WA. Mechanically Unfolded Titin Immunoglobulin Domains Refold Faster and More Accurately in Presence of Chaperone Alpha-B-Crystallin. Biophys J. 2017; 112(3):42a.
102. Kotter S, Unger A, Hamdani N, Lang P, Vorgerd M, et al. Human myocytes are protected from titin aggregation-induced stiffening by small heat shock proteins. J Cell Biol. 2014; 204(2):187– 202. [PubMed: 24421331]
103. Bullard B, Ferguson C, Minajeva A, Leake MC, Gautel M, et al. Association of the Chaperone αB-crystallin with Titin in Heart Muscle. J Biol Chem. 2004; 279(9):7917–24. [PubMed: 14676215]
104. Brinkmeier H, Ohlendieck K. Chaperoning heat shock proteins: Proteomic analysis and relevance for normal and dystrophin-deficient muscle. PROTEOMICS – Clin Appl. 2014; 8(11–12):875– 95. [PubMed: 24895218]
105. Zhu Y, Bogomolovas J, Labeit S, Granzier H. Single molecule force spectroscopy of the cardiac titin N2B element: effects of the molecular chaperone alphaB-crystallin with disease-causing mutations. J Biol Chem. 2009; 284(20):13914–23. [PubMed: 19282282]
106. Roach NT, Venkadesan M, Rainbow MJ, Lieberman DE. Elastic energy storage in the shoulder and the evolution of high-speed throwing in Homo. Nature. 2013; 498(7455):483–86. [PubMed: 23803849]
107. Minajeva A, Neagoe C, Kulke M, Linke WA. Titin-based contribution to shortening velocity of rabbit skeletal myofibrils. J Physiol. 2002; 540(1):177–88. [PubMed: 11927678]
108. Opitz CA, Kulke M, Leake MC, Neagoe C, Hinssen H, et al. Damped elastic recoil of the titin spring in myofibrils of human myocardium. Proc Natl Acad Sci. 2003; 100(22):12688–93. [PubMed: 14563922]
109. Dugdale TM, Dagastine R, Chiovitti A, Mulvaney P, Wetherbee R. Single Adhesive Nanofibers from a Live Diatom Have the Signature Fingerprint of Modular Proteins. Biophys J. 2005; 89(6): 4252–60. [PubMed: 16169972]
110. Sarkar A, Caamano S, Fernandez JM. The Mechanical Fingerprint of a Parallel Polyprotein Dimer. Biophys J. 2007; 92(4):L36–38. [PubMed: 17158577]
111. Bianco P, Reconditi M, Piazzesi G, Lombardi V. Is muscle powered by springs or motors? J Muscle Res Cell Motil. 2016:1–3. [PubMed: 26842420]
112. Caremani M, Pinzauti F, Reconditi M, Piazzesi G, Stienen GJM, et al. Size and speed of the working stroke of cardiac myosin in situ. Proc Natl Acad Sci. 2016; 113(13):3675–80. [PubMed: 26984499]
113. Takano M, Terada TP, Sasai M. Unidirectional Brownian motion observed in an in silico single molecule experiment of an actomyosin motor. Proc Natl Acad Sci. 2010; 107(17):7769–74. [PubMed: 20385833]
114. Freikamp A, Cost A-L, Grashoff C. The Piconewton Force Awakens: Quantifying Mechanics in Cells. Trends Cell Biol. 2016; 26(11):838–47. [PubMed: 27544876]
115. Meng F, Sachs F. Orientation-based FRET sensor for real-time imaging of cellular forces. J Cell Sci. 2012; 125(3):743–50. [PubMed: 22389408]
116. Austen K, Ringer P, Mehlich A, Chrostek-Grashoff A, Kluger C, et al. Extracellular rigidity sensing by talin isoform-specific mechanical linkages. Nat Cell Biol. 2015; 17(12):1597–1606. [PubMed: 26523364]
117. Grashoff C, Hoffman BD, Brenner MD, Zhou R, Parsons M, et al. Measuring mechanical tension across vinculin reveals regulation of focal adhesion dynamics. Nature. 2010; 466(7303):263–66. [PubMed: 20613844]
118. Arsenovic PT, Ramachandran I, Bathula K, Zhu R, Narang JD, et al. Nesprin-2G, a Component of the Nuclear LINC Complex, Is Subject to Myosin-Dependent Tension. Biophys J. 2016; 110(1): 34–43. [PubMed: 26745407]
119. Brenner MD, Zhou R, Conway DE, Lanzano L, Gratton E, et al. Spider Silk Peptide Is a Compact, Linear Nanospring Ideal for Intracellular Tension Sensing. Nano Lett. 2016; 16(3): 2096–2102. [PubMed: 26824190]
120. Prosser BL, Ward CW, Lederer WJ. X-ROS Signaling: Rapid Mechano-Chemo Transduction in Heart. Science. 2011; 333(6048):1440–45. [PubMed: 21903813]
121. Rivas-Pardo JA, Mártonfalvi Z, Manteca A, Eckels EC, Echelman DJ, et al. A Multi-Tool Mouse Model to Study the Elasticity of Native Titin. Biophys J. 2017; 112(3):167a.
122. Kohl J, Ng J, Cachero S, Ciabatti E, Dolan M-J, et al. Ultrafast tissue staining with chemical tags. Proc Natl Acad Sci. 2014; 111(36):E3805–14. [PubMed: 25157152]
123. Stagge F, Mitronova GY, Belov VN, Wurm CA, Jakobs S. Snap-, CLIP- and Halo-Tag Labelling of Budding Yeast Cells. PLoS ONE. 2013; 8(10):e78745. [PubMed: 24205303]
124. Horowits R, Kempner ES, Bisher ME, Podolsky RJ. A physiological role for titin and nebulin in skeletal muscle. Nature. 1986; 323(6084):160–64. [PubMed: 3755803]
125. Higuchi H. Changes in Contractile Properties with Selective Digestion of Connectin (Titin) in Skinned Fibers of Frog Skeletal Muscle. J Biochem (Tokyo). 1992; 111(3):291–95. [PubMed: 1587789]
126. Dobbie I, Linari M, Piazzesi G, Reconditi M, Koubassova N, et al. Elastic bending and active tilting of myosin heads during muscle contraction. Nature. 1998; 396(6709):383–87. [PubMed: 9845077]
127. Piazzesi G, Reconditi M, Linari M, Lucii L, Sun Y-B, et al. Mechanism of force generation by myosin heads in skeletal muscle. Nature. 2002; 415(6872):659–62. [PubMed: 11832949]
128. Reconditi M, Linari M, Lucii L, Stewart A, Sun Y-B, et al. The myosin motor in muscle generates a smaller and slower working stroke at higher load. Nature. 2004; 428(6982):578–81. [PubMed: 15058307]
129. Fenn WO. A quantitative comparison between the energy liberated and the work performed by the isolated sartorius muscle of the frog. J Physiol. 1923; 58(2–3):175–203. [PubMed: 16993652]

“筋収縮における主要な駆動力としてのタイチン蛋白のフォールディングの働き” への1件の返信